T.C.
SELÇUK ÜNĠVERSĠTESĠ SAĞLIK BĠLĠMLERĠ ENSTĠTÜSÜ
TÜRKĠYE’DE BULUNAN BAZI SIĞIR IRKLARININ
DGAT1 VE PRNP GEN POLĠMORFĠZMĠNĠN ARAġTIRILMASI
Ġclal ġAHĠN
YÜKSEK LĠSANS TEZĠ
BĠYOKĠMYA (VET.) ANABĠLĠM DALI
DanıĢman
Prof. Dr. Mehmet NĠZAMLIOĞLU
T.C.
SELÇUK ÜNĠVERSĠTESĠ SAĞLIK BĠLĠMLERĠ ENSTĠTÜSÜ
TÜRKĠYE’DE BULUNAN BAZI SIĞIR IRKLARININ
DGAT1 VE PRNP GEN POLĠMORFĠZMĠNĠN ARAġTIRILMASI
Ġclal ġAHĠN
YÜKSEK LĠSANS TEZĠ
BĠYOKĠMYA (VET.)ANABĠLĠM DALI
DanıĢman
Prof. Dr. Mehmet NĠZAMLIOĞLU
Bu araĢtırma Selçuk Üniversitesi Bilimsel AraĢtırma Projeleri Koordinatörlüğü tarafından 09202016 proje numarası ile desteklenmiĢtir.
i
ii. ÖNSÖZ
Hayvan yetiĢtiriciliği içerisinde sığır yetiĢtiriciliğinin, süt üretiminin tamamına yakınını ve et üretiminin de yaklaĢık 1/4’i karĢılanması nedeniyle önemi gittikçe artmaktadır. Sığırcılığın et ve süt üretiminde bu kadar büyük yer kapsaması, sığırın birçok biyolojik avantaja sahip olmasından ileri gelmektedir. Bu avantajlardan kısaca bahsetmek gerekirse; öncelikle sığıcılığın Türkiye için önemini, sığırın et ve süt üretimindeki yerini ortaya koymak gerekir. Sığır kaba yemleri hayvansal proteine dönüĢtürebilme yeteneği ve farklı koĢullara uyum sağlayabilmesi yönünden yetiĢtirici açısından büyük bir avantaj sağlamaktadır. Hayvan türleri içerisinde sığır, süt ve et verimi en yüksek olan hayvandır. Sığır aynı zamanda genetik ıslah çalıĢmalarına da yüksek düzeyde reaksiyon gösteren bir türdür. Bu yüzden sığır yetiĢtiriciliği, ülkemizin temel gıda ihtiyaçlarını karĢılayan ve yetiĢtiricilerimize ekonomik katkı sağlayan bir sektördür.
Bilinçli hayvan yetiĢtiriciliğinde en önemli unsur seleksiyondur. Bu amaçla süt hayvancılığını iyi düzeye çıkarmak için çevre ve yetiĢtirme Ģartlarının yanında seleksiyonunda dikkate alınması gerekmektedir.
Ġstenilen üstün özellikler ve bu özelliklerin yavrulara aktarılması da yetiĢtiriciliğin vazgeçilmez kuralıdır.
Islah çalıĢmalarında, istenilen özelliğe sahip erkek ve diĢi hayvanların seçimi ve damızlık değerinin bilinmesi önem taĢımaktadır. Islah çalıĢmaları, hayvanların verim özelliklerini artırıp ve bu yönde olan hayvanların iĢletmeye kazandırılması ve gelecek nesillere taĢınması açısından önemlidir.
Sunulan bu tezde, Türkiye’de bulunan bazı yerli sığır ırklarının süt yağı ve protein miktarı üzerinde etkili olan DGAT1 (diasilgliserol-asiltransferaz 1) K232A polimorfizminin Polimeraz Zincir Reaksiyonu (PZR) ve RFLP (Retriksiyon Enzimleri Uzunluk Polimorfizmi) ve PRNP (prion protein) geni promotor bölgesi 23 bç, intron 1 bölgesi 12 bç indel polimorfizmlerinin PZR yöntemleri kullanılarak ortaya konulması, bu sonuçların hayvan yetiĢtiriciliği ve ıslah çalıĢmalarında süt verimi yönünden seleksiyona katkı sağlaması amaçlanmıĢtır.
ii iii. ĠÇĠNDEKĠLER Sayfa SĠMGELER VE KISALTMALAR ... iv 1. GĠRĠġ ... 1 1.1. Aday Gen ... 1 1.2. DGAT1 Geni ... 1 1.3. DGAT Enzimi ... 2
1.4. DGAT1 Geni K232A Polimorfizmi... 3
1.5. DGAT1 K232A Polimorfizmi ile Ġlgili Yapılan ÇalıĢmalar ... 5
1.6. Prion Proteini (PrP) ... 6
1.7. Prion Protein Geni ... 7
1.8. BSE’ye Yatkınlık ... 8
1.9. PRNP Geni ile Ġlgili ÇalıĢmalar ... 8
1.11. ÇalıĢmaya Konu Olan Türkiye Yerli Sığır Irkları ... 9
1.10.1. Yerli Kara (YK) ... 9
1.10.2. Doğu Anadolu Kırmızısı (DAK)... 10
1.10.3. Boz Irk (BI) ... 11
1.10.4. Güney Anadolu Kırmızısı (GAK) ... 11
iii
2. GEREÇ VE YÖNTEM ... 14
2.1. Örneklerin Seçimi ... 14
2.2. DNA Ġzolasyonu ... 14
2.3. DNA Örneklerinin Miktar ve Kalitesinin Ölçülmesi ... 15
2.4. Polimeraz Zincir Reaksiyonu (PZR) ... 15
2.5. Agaroz Jel Elektroforez Analizi ... 16
2.6. PRNP geninin Genotiplenmesi ... 17
2.7. DGAT1 PZR-RFLP analizi ... 17
2.8. Ġstatistiksel Analizler... 18
3. BULGULAR ... 19
3.1. DGAT1 K232A Polimorfizmi Bulguları ... 19
3.1.1. DGAT1 Geni PZR Bulguları ... 19
3.1.2. PZR Ürünlerinin Restriksiyon Enzimi ile Kesimesi ... 19
3.1.3. DGAT1 Genotipi ... 20
3.1.4.Ġstatistiksel Analizlerin Sonuçları ... 21
3.2. PRNP Promotor 23 bç ve Ġntron 1 12 bç Ġndel Polimorfizmi ÇalıĢmasında Elde Edilen Bulgular ... 22
3.2.1. Prion Protein Geninin PZR Bulguları ... 22
3.2.2. PRNP Geninin Genotipi ... 23
iv
4. TARTIġMA ... 27
4.1. Bazı Yerli Sığır Irklarında PRNP Geni ... 29
4.2. Bazı Yerli Sığır Irklarında DGAT1 Geni ... 31
5. SONUÇ VE ÖNERĠLER ... 33
6. ÖZET ... 34
7. SUMMARY ... 35
8. KAYNAKLAR ... 36
v
iv. SĠMGELER VE KISALTMALAR
Ala Alanin
BAC Bacterial Artificial Chromosome (Bakteriyel Yapay Kromozomu)
bç Baz çifti
BI Boz Irk
BSE Bovine Spongiform Encephalopathy veya Deli Dana Hastalığı
BTA14 Bos Taurus chromosome 14, 14. kromozom çifti
CfrI Citrobacter freundii-I
cM Sentimorgan
DAG Diasilgliserol
DAK Doğu Anadolu Kırmızısı
DGAT Diasilgliserol-asiltransferaz
DGAT1 Diasilgliserol-asiltransferaz 1
ddMQ Di distile su
indel insersion-delesyon
GAK Güney Anadolu Kırmızısı
LD Linkaj Disequilibrum, BirleĢiklik Dengesizliği
Lys Lizin
MAG Monoasilgliserol
ng/ul Nanogram/ mikro litre
nmol Nanomol
PA Fosfotidik Asit
pmol Pikamol
PRNP Prion Protein Geni
PrP Prion Protein
PZR Polimeraz Zincir Reaksiyonu
QTL Quantitative Trait Loci, Kantitatif Özellik Lokusu RFLP Retriksiyon Enzimleri Uzunluk Polimorfizmleri
vi
TAG Triasilgliserol
TBE Tris-Borik Asit-EDTA
TSE Transmisible Spongiform Encephalopathy, BulaĢıcı Süngerimsi Beyin Hastalığı
YK Yerli Kara
1
1. GĠRĠġ
1.1. Aday Gen
Aday gen yaklaĢımları, kantitatif özellikleri etkileyen genlerin belirlenmesi ve haritalanmasını sağlayan bir yöntemdir. Aday bir gen, ilgili özelliğin fizyolojisini etkileyen biyolojik bir gen (fonksiyonel aday gen) veya fonksiyonel bir gen ile bağlantılı gen (pozisyonel aday gen) olarak tanımlanabilir. Süt proteinleri, somatotropik eksen (somatotropic axis) ve lenfosit antijenleri ile ilgili aday genler, sığırlarda en çok çalıĢılan genlerdir (Lacorte ve ark 2006). Örneğin: 20. sığır kromozomu (BTA20) üzerinde bulunan growth hormon reseptör genindeki bir polimorfizmin, süt verimine ve bileĢimine etkisi olduğuna dair güçlü bulgular bulunmuĢtur (Blott ve ark 2003). Tsiaras ve ark (2005), Holstein ineklerinde süt ve yağ verimini etkileyen Kappa-casein geninde bir polimorfizmi rapor etmiĢlerdir; Machado ve ark (2005), sığırlarında süt verimi ile BoLA DRB3 16 ve 29 allelleri arasında bir iliĢki bulmuĢlardır.
Mevcut sığır bileĢiklik haritasında (linkaj haritası) yeterli sayıda ve sıklıkta markör bulunmaktadır. Bu durum, kantitatif özellik lokuslarının (quantitative trait loci, QTL) tespitini ve QTL bölgelerinde aday genleri tanımlamayı amaçlayan genom tarama yaklaĢımlarının geliĢtirilmesini mümkün kılmıĢtır. Birçok genom tarama çalıĢması sonucunda süt verim özelliği üzerine önemli etkileri olan QTL’ler BTA6, 14, 20 ve 26 üzerinde bulunmuĢtur (Lacorte ve ark 2006).
1.2. DGAT1 Geni
Fine-mapping çalıĢmalarında 14. sığır kromozomu (BTA14)’ün proksimal ucunda, sütün yağ miktarını etkileyen bir QTL saptanmıĢtır (Spelman ve ark 2002, Grisart ve ark 2004, Kühn ve ark 2004, Pareek ve ark 2005, Strzalkowska ve ark 2005, Tantia ve ark 2006, Lacorte ve ark 2006, Nowacka-Woszuk ve ark 2008). Linkaj disequilibrum (LD) yöntemi, BULGE30 ve BULGE9 mikrosatelit markörleri ile sınırlandırılmıĢ 3.8 sentimorgan (cM)’lık aralıkta QTL’nin pozisyonunu belirlemek için kullanılmıĢtır. Bu aralığı kapsayan bakteriyel yapay kromozomu
2 (Bacterial Artificial Chromosome, BAC) klonları oluĢturulmuĢ ve bu bölgenin, güçlü bir aday gen olan diasilgliserol asil transferaz 1 (DGAT1) genini içerdiği gösterilmiĢtir (Grisart ve ark 2004).
Grisart ve ark (2002) ise DGAT1 geninin genomik ve cDNA sekanslarını karĢılaĢtırmıĢlar ve DGAT1 geninin 8.6 Kb’lık bir bölgeye yayıldığını göstermiĢlerdir. Sığır DGAT1 geninde 17 ekzon, 16 intron bulunmaktadır. 1. intron 3.6 Kb, 2. intron 1.9 Kb, kalan 14 intron ise yalnızca 92.4 bç uzunluğundadır. Sığır DGAT1 geninin transkripsiyon ürünü olan mRNA sekansı ise; 245 bç uzunluğunda 5’UTR, 1470 bp uzunluğunda protein kodlama bölgesive AATAAA poliadenilasyon sinyal kuyruğunu içeren 275 bp uzunluğundaki 3’UTR sekansından oluĢmaktadır. DGAT1 geninin transkripsiyonu sonucu oluĢan mRNA’nın translasyonu ile de 489 amino asit içeren (Grisart ve ark 2002), DGAT1 enzimi sentezlenmektedir (Spelman ve ark 2002, Thaller ve ark 2003, Grisart ve ark 2004, Kühn ve ark 2004, Casas ve ark 2005, Pareek ve ark 2005, Strzalkowska ve ark 2005, Tantia ve ark 2006, Lacorte ve ark 2006, Nowacka-Woszuk ve ark 2008, Näsland ve ark 2008).
ġekil 1.1 DGAT1 geninin anatomik yapısı (Grisart ve ark 2002).
1.3. DGAT1 Enzimi
AcylCoA:diacylglycecerol-acyltransferase 1 (DGAT1), hücresel gliserolipid metabolizmasında anahtar rol oynayan mikrosomal bir enzimdir. DGAT1 enzimi, diasilgliserol (DAG) ile fatty acyl coA’yı substrat olarak kullanır ve triasilgliserol (TAG) sentezinin son basamağını katalizler. TAG sentezinde kullanılan DAG: Gliserol-3-fosfat’tan sentezlenen fosfotidik asit (PA)’in hidroliz reaksiyonundan, monoasilgliserol (MAG)’ın esterifikasyonundan, triasilgliserol veya fosfolipid’lerin hidrolizinden gelmektedir (Cases ve ark 1998).
3
ġekil 1.2. TAG sentezinde DGAT1 enziminin iĢlevi(Shi ve Cheng 2009).
DGAT1 enzimi; bağırsaklardan yağ absorpsiyonunda, lipoprotein oluĢumunda, plazma triasilgliserol konsantrasyonunun regülasyonunda, adiposit hücrelerinde yağ depolanmasında, kaslarda enerji metabolizmasında ve süt veriminde rol oynamaktadır (Cases ve ark 1998).
1.4. DGAT1 Geni K232A Polimorfizmi
DGAT1 geninin 8. ekzonunda 10433 ve 10434 pozisyonunda bulunan adenin / adenin nükleotidlerinin, guanin / sitozin nükleotidlerine değiĢtiği bir mutasyon belirlenmiĢtir (Grisart ve ark 2002, Spelman ve ark 2002, Thaller ve ark 2003, Grisart ve ark 2004, Pareek ve ark 2005, Strzalkowska ve ark 2005, Lacorte ve ark 2006, Tantia ve ark 2006, Nowacka-Woszuk 2008, Näsland ve ark 2008, Signorelli ve ark 2009, Scotti ve ark 2010).
Monoasilgliserol
yolu
Gliserol fosfat
yolu
Monoasilgliserol
Diasilgliserol 1
4 10200 acagtgggct ccgtgctggc cctgatggtc tacaccatcc tcttcctcaa gctgttctcc 10260 taccgggacg tcaacctctg gtgccgagag cgcagggctg gggccaaggc caaggctggt 10320 gagggctgcc tcgggctggg gccactgggc tgccacttgc ctcgggaccg gcaggggctc 10380 ggctcacccc cgacccgccc cctgccgctt gctcgtagct ttggcaggta aggcggccaa 10440 cgggggagct gcccagcgca ccgtgagcta ccccgacaac ctgacctacc gcggtgagga 10500 tcctgccggg ggctgggggg actgcccggc ggcctggcct gctagccccg ccctcccttc 10560 cagatctcta ctacttcctc ttcgccccca ccctgtgcta cgagctcaac ttcccccgct 10620 ccccccgcat ccgaaagcgc ttcctgctgc ggcgactcct ggagatggtg aggcggggcc
ġekil 1.3. DGAT1 geninin 10200 – 10680 nükleotidleri arasındaki nükleotid dizisi ve 10433 ve 10434 posizyonundaki guanin/sitozin nükleotidleri (ebi.ac.uk 2009).
Bu mutasyon sonucunda DGAT1 geninin ürünü olan DGAT1 enziminin 232. pozisyonunda bulunan lizin (K) amino asidi alanin (A) amino asidine dönüĢtüğü belirlenmiĢtir (K232A polimorfizmi) (Spelman ve ark 2002, Thaller ve ark 2003, Grisart ve ark 2004, Kühn ve ark 2004, Casas ve ark 2005, Pareek ve ark 2005, Lacorte ve ark 2006, Tantia ve ark 2006, Nowacka-Woszuk 2008, Signorelli ve ark 2009, Scotti ve ark 2010). MGDRGGAGGSRRRRTGSRPSIQGGSGPAAAEEEVRDVGAGGDAPVRDTDKDGDVDVG SGHWDLRCHRLQDSLFSSDSGFSNYRGILNWCVVMLILSNARLFLENLIKYGILVDP IQVVSLFLKDPYSWPALCLVIVANIFAVAAFQVEKRLAVGALTEQAGLLLHGVNLAT ILCFPAAVAFLLESITPVGSVLALMVYTILFLKLFSYRDVNLWCRERRAGAKAKAAL AGKAANGGAAQRTVSYPDNLTYRDLYYFLFAPTLCYELNFPRSPRIRKRFLLRRLLE MLFLTQLQVGLIQQWMVPAIQNSMKPFKDMDYSRIVERLLKLAVPNHLIWLIFFYWL FHSCLNAVAELMQFGDREFYRDWWNSESITYFWQNWNIPVHKWCIRHFYKPMLRRGS SKWAARTAVFLASAFFHEYLVSIPLRMFRLWAFTGMMAQIPLAWIVGRFFRGNYGNA AVWLSLIIGQPVAVLMYVHDYYVLNREAPAAGT
ġekil 1.4. DGAT1 enziminin amino asit sekansı ve 232. pozisyondaki lizin amino asiti (uniprot.org 2009).
5
1.5. DGAT1 K232A Polimorfizmi ile Ġlgili Yapılan ÇalıĢmalar
BTA14’ün sentromerik bölgesinde sütün yağ içeriği ile ilgili olan kantitatif özellik lokusu (QTL) belirlenmiĢtir (Strzalkowska ve ark 2005, Nowacka-Woszuk ve ark 2008). Bu bölgede birbirinden bağımsız olarak Winter ve ark (2002) ve Grisart ve ark (2002) tarafından DGAT1 geni, süt verim özelliği bakımından güçlü bir aday gen olarak belirlenmiĢtir. Winter ve ark (2002), sütün yağ miktarındaki varyasyon ile ilgili 14. sığır kromozomu üzerindeki QTL bölgesiyle DGAT1’in eĢleĢtiğini göstermiĢtir.
Winter ve ark (2002)’dan bağımsız olarak Grisart ve ark (2002), 14. sğır kromozomu’nun sentromerik ucununda süt bileĢimine -özelliklede sütteki yağ miktarına- büyük etkisi olan bir QTL tespit etmiĢlerdir. Sütün yağ miktarına ve diğer süt karakterleri üzerinde büyük etkiye sahip olan DGAT1 geninin güçlü bir aday gen olduğunu ve DGAT1 geni üzerindeki K232A polimorfizmini göstermiĢlerdir. Trigliserid sentezinde anahtar rol oynayan DGAT1 enzimini kodlayan DGAT1 geni (Cases ve ark 1998, Kühn ve ark 2004, Casas ve ark 2005, Tantia ve ark 2006, Nowacka-Woszuk ve ark 2008, Näslund ve ark 2008) 14. sığır kromozomu üzerinde bulunmaktadır (Signorelli ve ark 2009).
Süt özelliği üzerine DGAT1 polimorfizminin etkisi, Spelman ve ark 2002, Kühn ve ark 2004, Näsland ve ark 2008, Pareek ve ark 2005 tarafından ayrıca doğrulanmıĢtır. Ayrshires sığır ırkı için sadece süt verimi, Holstein-Friesian ve Jersey için süt yağı, süt proteini ve süt verimi bakımından önemli sonuçlar belirlenmiĢtir. Allel değiĢimlerinin etkileri ortalama olarak proteine 2-3 kg, süte 20-30 kg’dır. Sütteki yağ verimine allel değiĢiminin ortalama etkisi Holstein-Friesian için 6 kg, Jersey için 3 kg’dır. Weller ve ark (2003), Ġsrail Holstein populasyonunda DGAT1 K232A polimorfizminin süt verimi, yağ verimi, sütteki yağ ve protein miktarıyla ilgili olduğunu bulmuĢlardır. Thaller ve ark (2003), DGAT1 K232A polimorfizminin sütteki yağ ve protein miktarına etkisinin yanında süt, süt yağı ve süt proteini verimine etkisini ve allel frekansını tahmin etmek amacıyla Fleckvich ve German Holstein granddaughter tasarımını kullanmıĢlardır. Lizin kodlayan varyantta gen polimorfizminin etkisi her ırk için ortalama olarak yağ miktarına % 31 ve protein
6 miktarına % 6 olduğu tespit edilmiĢtir. Diğer taraftan, lizin varyantının negatif etkisi ilk laktasyondan üçüncü laktasyona Holstein’da 260 – 320 kg ve Fleckvich’de ise 242 – 180 kg olarak açıklanmıĢtır.
Alanin allelinin süt veriminde artıĢla ilgili olmasına karĢın lizin kodlayan varyant sütteki yağ ve protein miktarının yanı sıra sütteki yağ veriminde artıĢla ilgili olduğu gözlenmiĢtir. Lizin varyantın en önemli etkisi, sütün yağ miktarının artıĢında olduğu görülmüĢtür (Strzalkowska ve ark 2005). Ayrıca Kaupe ve ark (2007), doğurganlık oranı (maternal non-return rate) ve dolayısıyla doğurgan dönemin uzunluğu üzerinde, DGAT1 geninin K allelinin (Lizin varyant) negatif etkiye sahip olabileceğini göstermiĢlerdir. Özellikle alanin alleline (A allel) nazaran lizin kodlayan allelin (K allel) sütteki yağ miktarının artıĢı ile ilgili olduğu saptanmıĢtır (Grisart ve ark 2002). Söz konusu polimorfizm sütün yağ, protein içeriği ve yağ verimiyle ilgili olan Leptin ve DGAT1 genlerinin genotipilerinin kombine olduğunu gösteren Kaminski ve ark (2006) tarafından 16 SNP (tek nükleotid polimorfizmi) içeren sığır mikroarray ile analiz edilmiĢtir.
Szyda ve Komisarek (2007), süt verim özelliğine etkisi olan BTN1A1, LEP, LEPR ve DGAT1 genlerinde 9 SNP analiz etmiĢlerdir. DGAT1 genindeki K232A polimorfizminin diğer polimorfizmlere göre sütteki yağ ve protein verimi üzerine etkisinin daha geniĢ olduğunu göstermiĢlerdir. Ayrıca K allelin insan sağlığı üzerinde negatif etkiye sahip olan sütteki doymamıĢ yağ asidi payında azalma ve doymuĢ yağ asidi payında artıĢ ile ilgili olduğu, ek olarak K allelin Charolaise ve Holstein sığırlarında kasın intramusküler yağ miktarı üzerinde pozitif etkiye sahip olduğu da tespit edilmiĢtir (Nowacka-Woszuk 2008). Casas ve ark (2005)’da Zebu sığırlarında, karkas ağırlığı ve K232A polimorfizmi arasında önemli bir ilgi bulunmadığını rapor etmiĢlerdir.
1.6. Prion Proteini (PrP)
Prion proteini (PrP), memelilerde TSE’nin (Transmisible Spongiform Encephalopathy) çeĢitli formlarıyla ilgilidir. Metabolik süreçlerde önemli rolü olduğu kanıtlanan prion proteininin amino asit sekansı, farklı memeli türlerinde benzerlik
7 gösterir. PrP’nin normal sinaptik fonksiyon için gerekli olduğu, hücreler arası haberleĢmeyi sağladığı ve anti-apoptotik sinyal molekülü olarak hareket ettiği sanılmaktadır (Czarnik ve ark 2007b).
1.7. Prion Protein Geni
PrP, prion protein geni (PRNP) tarafından kodlanır. PRNP, türlere bağlı olarak 2 veya 3 ekzon içerir (Czarnik ve ark 2007b). Sığır PRNP geni, kromozom BTA13 q17 bulunur ((Czarnik ve ark 2007a, Czarnik ve ark 2007b, (Czarnik ve ark 2009). Sander ve ark (2004), PRNP geni üzerindeki polimorfizmleri saptamak amacıyla sığır PRNP geninin 9350 bç uzunluğundaki kısmını sekanslamıĢlardır. PRNP geninin fonksiyonel olarak önemli iki bölgesi bulunmaktadır. Birinci bölge, promotor bölgesi olduğu farz edilen bölge, ekzon 1 ve ekzon 2’den oluĢmaktadır ve 5200 bç uzunluğundadır. Transkripsiyon baĢlama bölgesinin yaklaĢık olarak 2,5 kb upstreamındaki bölge tekrar sekansı içermediğinden promotor bölgesi olarak kabul edilmiĢtir. Ġkinci bölge ise ekzon 3 ve 795 bç uzunluğundaki PRNP açık okuma çerçevesinden meydana gelmektedir ve 4150 bç uzunluğundadır.
ġekil 1.5. Sander ve ark (2004) tarafından PRNP geni üzerindeki belirlenen 60 polimorfik bölge (a) ve PRNP geninin anatomik yapısı (b).
Sander ve ark (2004), PRNP geni üzerinde 36 polimorfizm belirlemiĢlerdir. Bunlardan 28’i tek nükleotid polimorfizmi (SNP, single nükleotide polymorphism), 3’ü iki nükleotid polimorfizmi, 3’ü tek baz insersiyon/delesyon (indel), 1’i iki baz
8 indel ve 1’i de 23 baz indel polimorfizmidir. Bu polimorfizmlerden hiç biri Prion proteininin amino asit sekansını etkilememektedir ayrıca sadece BSE’li hayvanlarda bulunan herhangi bir mutasyon gözlenmemiĢtir.
1.8. BSE’ye Yatkınlık
Sander ve ark (2004), PRNP geni promotor bölgesindeki 23 bç, intron 1 bölgesindeki 12 bç, 3’ UTR (3’ untranslated region) bölgesindeki 14 bç indel polimorfimlerin BSE ile ilgili olup olmadıklarını araĢtırmıĢlardır. Bu amaçla herbir polimorfik bölgenin allel ve genotipik frekanslarının, sağlıklı ve BSE’li sığırlar arasındaki dağılımında, herhangibi farklılık olup olmadığını araĢtırmıĢlardır. Sağlıklı ve BSE’li sığırlar arasında promotor 23 bç indel ve intron 1 12 bç indel polimorfizmi genotipik frekansı istatistiksel olarak önemli farklılıklar gözlenmiĢtir. Ancak bu farklılıklar, sağlıklı ve BSE’li sığırları tam olarak ayırabilecek bir neden değildir (Sander ve ark 2004).
1.9. PRNP Geni ile Ġlgili ÇalıĢmalar
Sığır PRNP geninin genomik yapısı ve DNA dizisi, sığır PRNP geni ve BSE’ye yatkınlık arasındaki iliĢki kadar fonksiyonunu da değerlendirmek için yararlı olacaktır. Mevcut çalıĢmalar ile sığır PRNP cDNA ve sığır PRNP geninin genomik yapısı tanımlanmıĢtır (Horiuchi ve ark 1998, Czarnik ve ark 2007b).
PRNP geni üzerinde ilk olarak açık okuma çerçevesi (open reading frame) 24 bç insersiyon-delesyon ve 3. ekzonun kırılma fragmentindeki (flanking fragment) değiĢme saptanmıĢtır (Goldmann ve ark 1991). PRNP geninde 388 mutasyon tanımlanmıĢtır, bu mutasyonların 37 tanesi delesyon insersiyon tipindedir. Özellikle PRNP geninin intron 1 bölgesindeki 12 bç insersiyon-delesyon (indel) ve promotor bölgesindeki 23 bç insersiyon-delesyon (indel) polimorfizmi ile ilgili çalıĢmalarda, BSE’nin genetik etiyolojisi tanımlanmaya çalıĢılmıĢtır (Czarnik ve ark 2007b). Sander ve ark 2005, Juling ve ark 2006, sağlıklı hayvan gruplarına göre BSE’li hayvan gruplarında 23 bç ve 12 bç delesyonlu allellerin daha çok bulunduğu saptamıĢlardır.
9 Daha ileriki çalıĢmalarda (Sander ve ark 2005, Juling ve ark 2006, Haase ve ark 2007), PRNP geninin transkripsiyonu ile PRNP promotor ve intron 1 haplotip varyantları arasında bir iliĢki saptanmıĢtır. 23 bç ve 12 bç delesyon haplotipine sahip hayvanlarda, heriki polimorfik lokusta insersiyon haplotipli hayvanlara göre daha yüksek PRNP geni ekspresyon seviyesi gözlenmiĢtir.
Seabury ve ark (2004), Nakamitsu ve ark (2006), Juling ve ark (2006), tarafından yapılan çalıĢmalar ile promotor ve intron 1 bölgesinde insersiyon-delesyon (indel) saptanan populasyonun genetik yapısındaki farklılıklar doğrulanmıĢtır. Süt performans özelliğini iyileĢtirmek için seçilen sığırlarda, PRNP geninin heriki polimorfik lokusu incelenmiĢ ve BSE’ye yatkınlık ile iliĢkiyi gösteren del/del genotipinin en yüksek frekansı, düĢük verim değerine ve yüksek doğal adaptasyon kabiliyetine sahip hayvan gruplarında kaydedilmiĢtir (Walawski 1999).
Czarnik ve ark (2007a), meme bezlerindeki fonksiyonel değiĢiklerde markör olarak kullanılabilen PRNP geninin promotor (23 bç indel) ve intron 1 (12 bp indel) üzerindeki indel polimorfizmlerini tanımlamıĢlardır. PRNP gen polimorfizmi ile süt yağı verimi ve sütteki protein miktarı arasında önemli bir korelasyon olduğunu göstermiĢlerdir. Aynı zamanda sütteki yağ ve protein miktarına PRNP promotor bölgesi (23 bç insersiyon) genotipinin etkisini tespit etmiĢlerdir.
1.10. ÇalıĢmaya Konu Olan Türkiye Yerli Sığır Irkları
1.10.1. Yerli Kara (YK)
Anadolu'nun otokton (kendiliğinden oluĢan) bir ırkıdır. Rengi siyah ve beden yapısı ufaktır. Türkiye sığır ırklarının ufak yapılısı ve Cidago yüksekliği (omuzbaĢı = 110 cm.) en düĢük olanıdır (Batu 1962). Et verimi düĢük, süt verimi azdır. Ġyi besi tutmaz. Son derece dayanıklıdır (UlubaĢ ve Günay 2004).
10 Resim 1.1. Yerli Kara sığır ırkı (turkhaygen.gov.tr 2010).
1.10.2. Doğu Anadolu Kırmızısı (DAK)
Resim 1.2. Doğu Anadolu Kırmızısı (turkhaygen.gov.tr 2010).
Doğu ve Kuzey-Doğu Anadolu'nun bir ırkıdır. Rengi açık kırmızıdan koyu kestane rengine kadar değiĢir. Bazı hayvanlarda arka bacağın iç taraflarında beyaza kadar değiĢen açık renkler görülebilir. DAK Türkiye'nin en önemli sığır eti kaynağıdır (Batu 1962).
11
1.10.3. Boz Irk (BI)
Resim 1.3. Boz Irk sığırı (turkhaygen.gov.tr 2010).
Anadolu ve Trakya'nın yerli ırkıdır. Köken itibariyle Anadolu yerli ırkları arasında Bos taurus prigeminus grubuna (kemik ve kafatası yapısı ile belirgin, iri yapılı grup) giren tek ırktır. Rengi açık gümüĢten koyu kül rengine kadar değiĢir. Boğalarda göz etrafında siyah bir halka vardır (Batu 1962). En önemli özelliği, sağlam vücut yapısı ve iyi besi tutmasıdır (UlubaĢ ve Günay 2004).
1.10.4. Güney Anadolu Kırmızısı (GAK)
12 Torosların güneyinde kalan Akdeniz Bölgesi ile Güney-Doğu Anadolu Bölgesi bu ırkın yayılma alanıdır. Açık sarıdan koyu kırmızıya kadar değiĢen renk varyasyonları gösterir. En çok görülen tarçın rengidir. Sağrı (memeli hayvanlarda bel ile kuyruk arasındaki dolgun ve yuvarlakça bölüm), cidagodan 5 cm. kadar daha yüksek ve kuyruk bağlantısı yukarıdandır. Diğer yerli ırklara göre süt verimi ve et verimi bakımından en verimli olanıdır. Diğer ırklara göre geliĢmesi daha erkendir. Ġlk yavrularını üç yaĢtan itibaren verirler. Sağımının yapılabilmesi için buzağının emmesi veya buzağının ineğin yanında olması gerekir. Bu nedenle makinalı sağıma uygun değildir (Batu 1962).
1.10.5. Zavot (ZAV)
Resim 1.5. Zavot sığır ırkı (turkhaygen.gov.tr 2010)
Kars ilinin aynı isimle anılan bölgesinde yetiĢtirilen, yerli sığırlarla Kafkasya'dan getirilen Simental ve Ġsviçre Esmeri birleĢtirilmesiyle elde edilmiĢtir. Peynir yapımı ile uğraĢan mandıra ve civarında yetiĢtirilir (Batu 1962).
13 Bu çalıĢmada;
Türkiye’de bulunan bazı yerli sığır ırklarının DGAT1 K232A polimorfizminin PZR-RFLP tekniği kullanılarak ve PRNP promotor 23 bç ve intron 1 12 bç indel polimorfizmi PZR tekniği kullanılarak ortaya konulması amaçlanmıĢtır.
14
2. GEREÇ VE YÖNTEM
2.1. Örneklerin Seçimi
TÜRKHAYGEN-I projesi kapsamında örnekleme çalıĢması yapılan Boz Irk (BI), Yerli Kara (YK), Güney Anadolu Kırmızısı (GAK), Doğu Anadolu Kırmızısı (DAK) ve Zavot (ZAV) ırklarına ait toplma 122 adet kan örnekleri kullanıldı (Çizelge 2.1).
Çizelge 2.1. ÇalıĢmada kullanılan örneklerin sayısı.
Irk DiĢi (n) Erkek (n) Toplam (n)
GAK 14 13 27 YK 12 13 25 BI 13 13 26 DAK 15 15 30 ZAV 2 12 14 Toplam 56 66 122 2.2. DNA Ġzolasyonu
Kan örneklerinden standart fenol/kloroform yöntemi kullanılarak DNA izolasyonu yapıldı.
- Falkon tüplere 10 ml K3EDTA’lı kan örneği konuldu, üzerine Mg++ iyonlarını bağlayarak DNAaz aktivitesini engelleyip DNA’yı parçalanmaktan korumak için 0.5 ml 0,5 M edta (pH 8.0) ilave edilerek bu karıĢım 2X lysıs buffer ile 50 ml’ye tamamlandı.
- 10 dakika boyunca alt üst edilip 30 dakika buzun içinde bekletilen tüpler Buzdan alındıktan sonra 3000 rpm de +40C de 10 dakika santrifüj edildi. Santrifüj sonrası tüpün süpernatant fazı atıldı (tüpdeki tüm solüsyonlar döküldü).
- Tüpteki pellet üzerine 3 ml salt/edta eklenerek iyice vortexlendi. Daha sonra üzerine proteinleri denatüre edip proteazların iĢlevine hazır hale getirmek için 0,3 ml %10 luk SDS solüsyonu ve DNA’yı parçalayan stoplazmik enzimleri, DNA’yı bağlayan nüklear proteinleri yıkmak için 150 µl proteinaz K (10 mg/ml) eklenerek örnekler 55 0
15 - Bekleme süresi sonunda tüp üzerine 3 ml fenol (pH 8.0) eklendi. Tüpler 20 saniye oldukça sert bir Ģekilde çalkalandıktan sonra 5 dakika da yumuĢak bir Ģekilde ters düz edilip, 3000 rpm de +40C de 10 dakika santrifüj edildi. Santrifüj sonunda tüplerdeki süpernatant kısmı yeni steril tüplere alınıp, üzerine 240 µl fenol:kloroform:ızoamil alkol (25:24:1) eklendi. Tüpler 20 saniye oldukça sert bir Ģekilde çalkalandıktan sonra 5 dakika da yumuĢak Ģekilde ters düz edilerek, 3000 rpm de +40
C de 10 dakika santrifüj edildi. Santrifüj sonunda tüplerdeki süpernatant kısmı yeni tüplere alındı. - Süpernatın 1/10 kadar 3 M NaAc (pH 5.2), DNA’yı çökeltmek için alınan süpernant kısmının 2,5 katı kadar -200
C de soğutulmuĢ %95’lik etanol eklendi. Tüpler sert bir Ģekilde sallandı ve yoğunlaĢıp çöken DNA pipet ile alınarak yeni bir ependorf tüpe aktarıldı. Pellet kurutularak alkol uzaklaĢtırıldı. Pellet, ddMQ ile sulandırıldı.
- Sulandırılan DNA’lar -200C’de kullanılmak üzere saklandı.
2.3. DNA Örneklerinin Miktar ve Kalitesinin Ölçülmesi
Ġzole edilen DNA’lar Thermo scientific ND-1000 Nanodrop ile miktar ve kalitesi ölçüldü.
2.4. Polimeraz Zincir Reaksiyonu (PZR)
Elde edilen DNA’ların DGAT1 gen bölgesinin 411 baz çift (bç) lik fragmenti ve PRNP gen bölgesinin 47784-47883 ve 49686-49776 nükleotidleri arasında bulanan iki bölgesi primerler kullanılarak PZR yöntemi ile yükseltgendi. PZR protokolü olarak bir reaksiyonda 1x Mg++ free, PZR buffer (Fermentas), 200 mM dNTP (Fermentas), 1.5 mM MgCl++, 0.375 ünite Taq polimeraz (Fermentas), 5 pMol her bir primer çifti (Çizelge 2.2) ve 100 ng DNA template kullanıldı. PZR karıĢımı 15 µl olarak hazırlandı.
16
Çizelde 2.2. Kullanılan primer sekansları.
DGAT1 geni F 5’-CACCATCCTCTTCCTCAAG-3’
Lacorte ve ark 2006 R 5’-GGAAGCGCTTTCGGATG-3’ PRNP geni 47784-47883 gen bölgesi F 5’-GTGCCAGCCATGTAAGTG-3’ Sander ve ark 2004 R 5’-TGGACAGGCACAATGGG -3’ PRNP geni 49686-49776 gen bölgesi F 5’-TTACCCTCCTGGTTAGGAG -3’ Sander ve ark 2004 R 5’-CTAGATTCCTACACACCAC -3’
PZR, MJ Research PTC-200 thermal cycle ve touchdown PZR profili kullanılarak iki aĢamada gerçekleĢtirildi. PZR profili ise DGAT1 gen bölgesi için 95°C’de 4 dakika ile tam bir denatürasyon sonrası birinci aĢamada 16 döngü için 94°C’de 30 saniye denatürasyon, primerlerin ideal bağlanma noktasının sağlanması için 60°C’den baĢlayarak her bir döngüde 0.5°C düĢürülen ve 30 saniye süren annealing ve 72°C’de elongation sağlandı. Ġkinci aĢamada 94°C’de 30 saniye denatürasyon, 52°C’de 30 saniye annealing ve 72°C’de 1 dakika elongation olacak Ģekilde toplam 25 döngü kullanıldı. Son olarak örnekler 72°C’de 10 dakika tutularak tam bir adenilizasyon sağlandı.
PRNP gen bölgesinin 47784-47883 ve 49686-49776 nükleotidleri arasında bulanan iki bölgesi için PZR profili ise 95°C’de 4 dakika ile tam bir denatürasyon sonrası birinci aĢamada 16 döngü için 94°C’de 30 saniye denatürasyon, primerlerin ideal bağlanma noktasının sağlanması için 60°C’den baĢlayarak her bir döngüde 0.5°C düĢürülen ve 30 saniye süren annealing ve 72°C’de elongation sağlandı. Ġkinci aĢamada 94°C’de 30 saniye denatürasyon, 52°C’de 30 saniye annealing ve 72°C’de 30 saniye elongation olacak Ģekilde toplam 25 döngü kullanıldı. Son olarak örnekler 72°C’de 10 dakika tutularak tam bir adenilizasyon sağlandı.
2.5. Agaroz Jel Elektroforez Analizi
PZR iĢleminden sonra elde edilen PZR ürünlerini değerlendirmek amacıyla %2’li agaroz jel kullanıldı. DGAT1 geni PZR ürününden 5 µl alınıp 4µl 6x loading dye ile, Prion geni PZR ürünlerinden ise 15 µl alınıp 4µl 6x loading dye ile
17 karıĢtırılarak kuyucuklara yüklendi. Jel 40 dakika 150 voltluk elektrik akımına tabi tutuldu. Ayrıca allel boyutlarınının tahmini için 100 bç ladder kullanıldı. Jeller etidyum bromid’te 5-10 dk bekletilerek boyandı ve UV ıĢığı altında fotoğrafı çekilerek değerlendirildi.
2.6. PRNP Geninin Genotiplenmesi
PRNP geninin 47784-47883 (promotor) ve 49686-49776 (intron 1) gen bölgelerinin % 2’lik agaroz jel elektroforezinde ayrıĢtırılarak, UV ıĢığı altında fotoğrafı çekilip değerlendirilmesi sonucunda; promotor bölgesi için 91 ve 103 bç büyüklüğünde bantlar gözlendi. Genotipleme yapılırken sadece 91 bç büyüklüğünde bant görüldüğünde del/del homozigot; 91 ve 103 bç büyüklüğündeki bantlar birlikte görüldüğünde del/ins heterozigot; 103 bç büyüklüğündeki bantlar görüldüğünde ise ins/ins homozigot Ģeklinde genotiplendirilmesi değerlendirildi.
Ġntron 1 bölgesi için ise 100 ve 123 bç büyüklüğünde bantlar gözlendi. Genotipleme yapılırken sadece 100 bç büyüklüğünde bant görüldüğünde del/del homozigot; 100 ve 123 bç büyüklüğündeki bantlar birlikte görüldüğünde ins/del heterozigot; 123 bç büyüklüğündeki bantlar görüldüğünde ise ins/ins homozigot Ģeklinde genotiplendirilmesi değerlendirildi.
2.7. DGAT1 PZR-RFLP Analizi
Değerlendirme sonucunda geriye kalan 10µl PZR ürünlerinde RFLP analizi için restriksiyon endonükleaz (RE) kesimi yapıldı. Bu amaçla 10 µl PZR ürünü, 8 µl ddMQ, 5 U CfrI enzimi, 1 µl restriksiyon enzim kesim tamponu ile toplam 20 µl’lik restriksiyon enzim kesim karıĢımları hazırlandı. Tam bir RE kesimi için 37 °C’de 6-12 saat ve RE inaktivasyonu için etüvde 65°C’de 20 dakika bekletildi. RE kesim ürünleri % 2’lik agaroz jelde 40 dakika 150 voltta yürütüldü ve bantların büyüklüğünü tahmin etmek için 100 bç’lik ladder kullanıldı. Jeller etidyum bromid’te 5-10 dk bekletilerek boyandı ve UV ıĢığı altında fotoğrafı çekilip değerlendirildi.
18 Değerlendirme sonucunda, DGAT1 geni için 203, 208, 411 bç büyüklüğünde bantlar gözlendi. Genotipleme yapılırken sadece 203 ve 208 bç büyüklüğünde bant görüldüğünde AA (ala/ala) homozigot; 203 ve 208 bç büyüklüğündeki bantlar ile 411 bç büyüklüğündeki bant birlikte görüldüğünde AK (ala/lys) heterozigot; 411 bç büyüklüğündeki bantlar görüldüğünde ise KK (lys/lys) homozigot Ģeklinde genotiplendirilmesi değerlendirildi.
2.8. Ġstatistiksel Analizler
Verilerin istatistiksel analizi TFPGA, version 1.3 8 (Miller, 1997) programı ile yapıldı.Hardy-Weinberg dengesi tam olasılık testi (Haldane, 1954) ile değerlendirildi.
19
3. BULGULAR
3.1. DGAT1 K232A Polimorfizmi Bulguları
3.1.1. DGAT1 Geninin PZR Bulguları
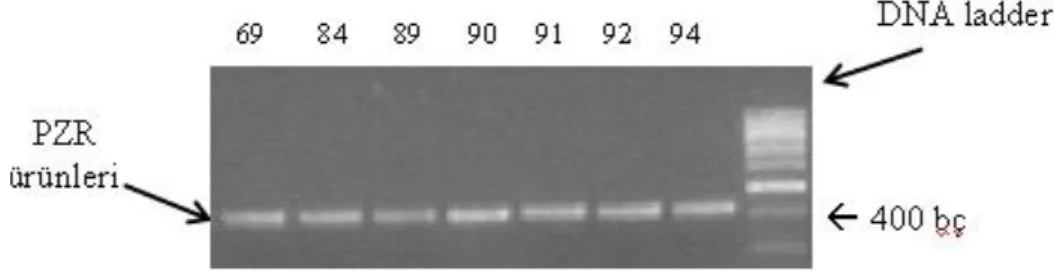
DGAT1 10230 – 10644 nükleotidleri arasında bulanan bölgesi, primerler kullanılarak PZR yöntemi ile yükseltgenmiĢ ve agaroz jel elektroforezinde yürütülüp UV ıĢığı altında çekilen jel fotoğraflarında 411 bç uzunluğunda gözlenen bantlar ġekil 3.1’de gösterilmiĢtir.
ġekil 3.1. DGAT1 geninin 10230 – 10644 nükleotidleri arasıdaki bölgesinin PZR ürününün, agaroz jel fotoğrafı.
3.1.2. PZR Ürünlerinin Restriksiyon Enzimi ile Kesilmesi
YükseltgenmiĢ DGAT1 geninde K232A polimorfizmi PZR-RFLP tekniğiyle belirlenmiĢtir. Agaroz jel elektroforezinde ayrıĢtırılan ve UV ıĢığı altında çekilen jel fotoğraflarında üç farklı sonuç gözlenmiĢtir. Bunlardan ilki, kesmenin gerçekleĢmediği 411 bç uzunluğundaki bant, ikincisi kesme iĢleminin gerçekleĢtiği 203 bç ve 208 bç uzunluğundaki bantlar, üçüncüsü ise 203 bç ve 208 bç ile 411 bç uzunluğundaki bantlar gözlenmiĢtir.
20
ġekil 3.2. DGAT1 geninin 10230 – 10644 gen bölgesinin PZR ürününün, CfrI restriksiyon enzimi ile kesim sonrası, agaroz jel fotoğrafı. 84, 90, 91, 92 nolu örnekler (203 ve 208 bç) Ala/Ala, 69 ve 89 nolu örnekler (203, 208 ve 411 bç) Ala/Lys, 94 nolu örnek (411 bç) Lys/Lys genotipini göstermektedir.
3.1.3. DGAT1 Genotipi
Yükselgenen DGAT1 gen bölgesinin 10230-10644 nükleotidleri arasında bulanan bölgesi CfrI enzimi ile kesilip, elde edilen enzim kesim ürünleri agaroz jel elektroforezinde yürütülmüĢ, UV ıĢığı altında fotoğrafı çekilip değerlendirilmiĢtir.
Değerlendirme sonucunda, DGAT1 geni için 203, 208, 411 bç büyüklüğünde bantlar gözlenmiĢtir. Genotipleme yapılırken sadece 203 ve 208 bç büyüklüğünde bant görüldüğünde AA (Ala/Ala) homozigot; 203 ve 208 bç büyüklüğündeki bantlarla 411 bç büyüklüğündeki bant birlikte görüldüğünde AK (Ala/Lys) heterozigot; 411 bç büyüklüğündeki bantlar görüldüğünde ise KK (Lys/Lys) homozigot Ģeklinde genotiplendirilmesi değerlendirilmiĢtir.
21
Çizelge 3.1. Boz Irk, Yerli Kara, Güney Anadolu Kırmızı, Doğu Anadolu Kırmızısı ve Zavot ırklarında gözlenen genotipler.
GAK 7 13 7 25,93 48,14 25,93 YK 8 13 4 32,00 52,00 16,00 BI 4 10 10 16,66 41,67 41,67 DAK 5 12 12 17,24 41,38 41,38 ZAV 4 6 4 28,57 42,86 28,57 23,52 45,37 31,09 Toplam 28 54 37 AK KK
Gözlenen Genotipler Gözlenen Genotipler %
AA AK KK AA
3.1.4. Ġstatistiksel Analizlerin Sonuçları
Genotiplemesi yapılan verilerin, allel ve genotip frekansları hesaplanmıĢ, χ2
testi ile değerlendirilmiĢtir. Anlamlılık seviyesi 0,05 alınmıĢtır.
Çizelge 3.2. DGAT1 K232A genotipik, allelik frekans, gözlenen, beklenen heterozigot değerleri.
GAK 0,25 0,50 0,25 1,00 0,50 0,50 48,15 50,00
YK 0,34 0,48 0,18 1,00 0,58 0,42 52,00 48.72
BI 0,14 0,47 0,38 0,66 0,38 0,62 41,60 46,80
DAK 0,14 0,47 0,38 0,66 0,38 0,62 41,60 46,80
ZAV 0,25 0,50 0,25 0,61 0,50 0,50 42,80 50,00
Genotipik Frekans Allelik Frekans KK
Heterozigot % A K Observed
(Ho)
1Hardy-Weinberg denge testi için Exact Probability (Haldene, 1954) * önemli ; EP < 0,05
AA AK EP1 Expected
(He)
DGAT1 K232A polimorfizmi için yapılan genotiplemede; 37 (% 31,09) hayvanda Lys/Lys genotipi, 54 (% 45,37) Lys/Ala genotipi, 28 (% 23,52) Ala/Ala genotipi bulundu. GAK, YK, BI, DAK ve ZAV sığır ırklarında Lys/Lys genotipi (sırasıyla % 25,93- %16 - % 41,67 - % 41,38 - % 28,57), Lys/Ala genotipi (sırası ile % 48,14 - % 52 - % 41,67 - % 41,38 - % 42,86), Ala/Ala genotipi (sırası ile % 25,93 - % 32 - % 16,66 - % 17,24 - % 28,57) olarak bulundu. Lys/Lys genotipi referans alınarak yapılan istatistiksel hesaplamada heterozigot Lys/Ala genotipinin BI, YK, GAK, DAK ve ZAV sığır ırkları arasındaki dağılımlarında istatistiki olarak anlamlı bir fark tespit edilemedi (χ2
22
3.2. PRNP Promotor 23 bç ve Ġntron 1 12 bç Ġndel Polimorfizmi ÇalıĢmasında Elde Edilen Bulgular
3.2.1. Prion Protein Geninin PZR Bulguları
Fenol/kloroform yöntemiyle izole edilen DNA’ların PRNP geninin 47784 – 47883 (promotor) ve 49686 – 49776 (intron 1) nükleotidleri arasında bulanan bölgesi, primerler kullanılarak PZR yöntemi ile yükseltgenmiĢ ve agaroz jel elektroforezinde ayrıĢtırılmıĢtır. UV ıĢığı altında çekilen jel fotoğraflarında intron 1 bölgesi için 91 ve 103 bç büyüklüğünde, promotor bölgesi için ise 100 ve 123 bç büyüklüğünde bantlar gözlenmiĢtir.
Yükseltgenen PRNP geninin 49686 – 49776 (intron 1) nükleotidleri arasında bulanan bölgesinin agaroz jel elektroforezinde yürütülüp, UV ıĢığı altında çekilen jel fotoğraflarında üç farklı sonuç gözlenmiĢtir. Bunlardan ilki, delesyonun gerçekleĢmediği 91 bç uzunluğundaki bant, ikincisi, delesyonun ve insersiyonun gerçekleĢtiği 91 bç ve 103 bç uzunluğundaki bantlar, üçüncüsü insersiyonun gerçekleĢtiği 103 bç uzunluğundaki bantlar gözlenmiĢtir.
Yükseltgenen PRNP geninin 47784 – 47883 (promotor) nükleotidleri arasında bulanan bölgesinin agaroz jel elektroforezinde yürütülüp, UV ıĢığı altında çekilen jel fotoğraflarında üç farklı sonuç gözlenmiĢtir. Bunlardan ilki, delesyonun gerçekleĢmediği 100 bç uzunluğundaki bant, ikincisi delesyonun ve insersiyonun gerçekleĢtiği 100 bç ve 123 bç uzunluğundaki bantlar, üçüncüsü insersiyonun gerçekleĢtiği 123 bç uzunluğundaki bantlar gözlenmiĢtir.
23
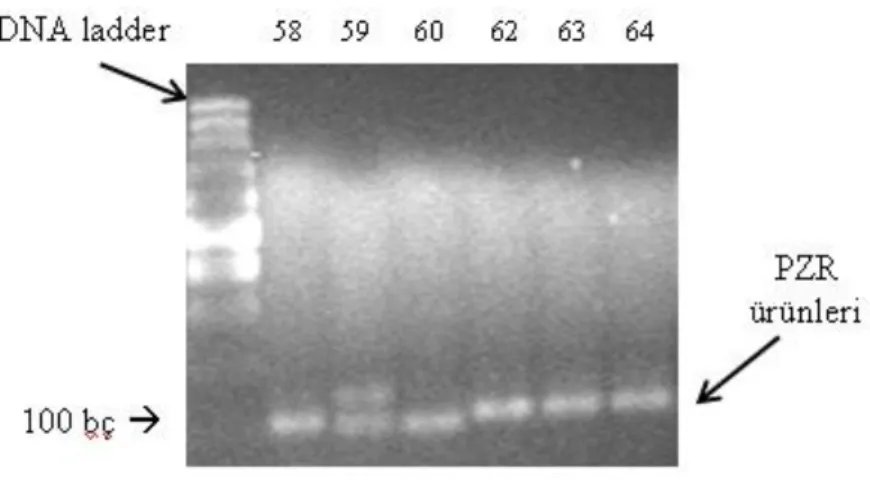
ġekil 3.3. PRNP geninin 49686 – 49776 (intron 1) nükleotidleri arasında bulanan bölgesinin PZR ürününün, agaroz jel fotoğrafı. 58 ve 60 tüp nolu örnekler (91 bç) del/del; 59 tüp nolu örnekler (91 ve 103 bç) del/ins; 62, 63 ve 64 tüp nolu örnekler (103 bç) ins/ins genotipini göstermektedir.
ġekil 3.4. PRNP geninin 49686 – 49776 (intron 1) nükleotidleri arasında bulanan bölgesinin PZR ürününün, agaroz jel fotoğrafı. 58 ve 60 nolu örnekler (91 bç) del/del; 59 nolu örnekler (91 ve 103 bç) del/ins; 62, 63 ve 64 nolu örnekler (103 bç) ins/ins genotipini göstermektedir
3.2.2. PRNP Geninin Genotipi
PRNP geninin 47784 – 47883 (promotor) ve 49686 – 49776 (intron 1) nükleotidleri arasında bulanan bölgesi PZR ile yükseltgenip, elektroforezinde yürütülmüĢ, UV ıĢığı altında fotoğrafı çekilip değerlendirilmiĢtir.
24 Değerlendirme sonucunda, intron 1 bölgesi için 91 ve 103 bç büyüklüğünde bantlar gözlenmiĢtir. Genotipleme yapılırken sadece 91 bç büyüklüğünde bant görüldüğünde (del/del) homozigot; 91 veya 103 bç büyüklüğündeki bantlar birlikte görüldüğünde (del/ins) heterozigot; sadece 103 bç büyüklüğündeki bantlar görüldüğünde ise (ins/ins) homozigot Ģeklinde genotiplendirilmesi
değerlendirilmiĢtir.
Promotor bölgesi için 100 ve 123 bç büyüklüğünde bantlar gözlenmiĢtir. Genotipleme yapılırken sadece 100 bç büyüklüğünde bant görüldüğünde (del/del) homozigot; 100 veya 123 bç büyüklüğündeki bantlar birlikte görüldüğünde (del/ins) heterozigot; sadece 123 bç büyüklüğündeki bantlar görüldüğünde ise (ins/ins) homozigot Ģeklinde genotiplendirilmesi değerlendirilmiĢtir.
Çizelge 3.3. BI, YK, GAK, DAK ve ZAV ırklarında PRNP geninin intron 1 bölgesinde gözlenen genotipler. GAK 2,00 9,00 7,00 11,11 50,00 38,89 YK 3,00 8,00 14,00 12,00 32,00 56,00 BI 9,00 4,00 9,00 40,91 18,18 40,91 DAK 5,00 3,00 21,00 17,24 10,38 72,41 ZAV 0,00 6,00 7,00 0,00 46,15 53,85 del/ins ins/ins
Gözlenen Genotipler Gözlenen Genotipler %
del/del del/ins ins/ins del/del
54,20
Toplam 19,00 30,00 58,00 17,77 28,03
Çizelge 3.4. BI, YK, GAK, DAK ve ZAV ırklarında PRNP geninin promotor bölgesinde gözlenen genotipler. GAK 17,00 2,00 6,00 68,00 8,00 24,00 YK 13,00 4,00 5,00 59,09 18,18 22,73 BI 6,00 9,00 9,00 25,00 37,50 37,50 DAK 14,00 7,00 3,00 58,33 29,17 12,50 ZAV 3,00 3,00 5,00 27,27 27,27 45,46 50,00 23,58 26,41 Toplam 53,00 25,00 28,00 del/ins ins/ins
Gözlenen Genotipler Gözlenen Genotipler %
25
3.2.3. Ġstatistiksel Analizlerin Sonuçları
Genotiplemesi yapılan verilerin, allel ve genotip frekansları hesaplanmıĢ, χ2
testi ile değerlendirilmiĢtir. Anlamlılık seviyesi 0,05 alınmıĢtır.
Çizelge 3.5. PRNP intron 1 12 bp indel genotipik, allelik frekans, gözlenen, beklenen heterozigot değerleri. GAK 0,13 0,46 0,41 0,62 0,36 0,64 50,00 46,10 YK 0,08 0,40 0,52 0,33 0,28 0,72 32,00 40,30 BI 0,25 0,50 0,25 0,003* 0,50 0,50 18,10 50,00 DAK 0,05 0,34 0,61 0,0006* 0,22 0,78 10,30 34,70 ZAV 0,05 0,36 0,59 1,00 0,23 0,77 46,10 35,50
Genotipik Frekans Allelik Frekans ins/ins
Heterozigot % del ins Observed
(Ho)
1Hardy-Weinberg denge testi için Exact Probability (Haldene, 1954) * önemli ; EP < 0,05
del/del del/ins EP1 Expected
(He)
PRNP intron 1 12 bç indel polimorfizmi için yapılan genotiplemede; 58 (% 54,20) hayvanda ins/ins genotipi, 30 (% 28,3) ins/del genotipi, 19 (% 17,77) del/del genotipi bulundu. GAK, YK, BI, DAK ve ZAV sığır ırklarında ins/ins genotipi (sırasıyla % 38,89 - % 56 - % 40,91 - % 72,41 - % 53,85), ins/del genotipi (sırası ile % 50 – % 32 - % 18,18 - % 10,34 - % 46,15) del/del genotipi (sırası ile % 11,11 - % 12 - % 40,91 - % 17,24 - % 0) olarak bulundu. ins/ins genotipi referans alınarak yapılan istatistiksel hesaplamada heterozigot ins/del genotipinin GAK, YK, BI, DAK ve ZAV sığır ırkları arasındaki dağılımlarında istatistiki olarak anlamlı bir fark tespit edildi (χ2= 10,66). Bu farklılığın sebebinin ortaya konması için Çizelge 3.3’deki veriler kullanıldığında; ins/del genotipinin BI’da 4 (% 18,18) hayvanda, DAK’da 3 (% 10,34) hayvanda mevcut olduğu görülmektedir.
26
Çizelge 3.6. PRNP promotor 23 bp indel genotipik, allelik frekans, gözlenen ve beklenen heterozigot değerleri. GAK 0,52 0,40 0,08 0,0001* 0,72 0,28 8,00 40,30 YK 0,46 0,44 0,10 0,009* 0,68 0,32 18,10 43,40 BI 0,19 0,49 0,32 0,24 0,44 0,56 37,50 49,20 DAK 0,53 0,40 0,07 0,29 0,73 0,27 29,10 39,50 ZAV 0,17 0,48 0,35 0,21 0,41 0,59 27,20 48,40
1Hardy-Weinberg denge testi için Exact Probability (Haldene, 1954) * önemli ; EP < 0,05
del/del del/ins EP1 Expected
(He) Genotipik Frekans Allelik Frekans
ins/ins
Heterozigot % del ins Observed
(Ho)
PRNP promotor 23 bç indel polimorfizmi için yapılan genotiplemede; 28 (% 26,41) hayvanda ins/ins genotipi, 25 (% 23,58) ins/del genotipi, 53 (% 50) del/del genotipi bulundu. GAK, YK, BI, DAK ve ZAV sığır ırklarında ins/ins genotipi (sırasıyla % 24 - % 22,73 - % 37,50 - % 29,17 - % 27,27), ins/del genotipi (sırası ile % 8 – % 18,18 - % 37,50 - % 29,17 - % 27,27) del/del genotipi (sırası ile % 68 - % 59,09 - % 25 - % 58,33 - % 27,27) olarak bulundu. ins/ins genotipi referans alınarak yapılan istatistiksel hesaplamada heterozigot ins/del genotipinin GAK, YK, BI, DAK ve ZAV sığır ırkları arasındaki dağılımlarında istatistiki olarak anlamlı bir fark tespit edildi (χ2= 12). Bu farklılığın sebebinin ortaya konması için Çizelge 3.4’deki veriler kullanıldığında; ins/del genotipinin GAK sığır ırkında 2 (% 8), YK sığır ırkında 4 (% 18,18) hayvanda mevcut olduğu görülmektedir.
27
4. TARTIġMA
Sunulan çalıĢmada, Türkiye’de bulunan bazı yerli sığır ırklarının süt yağı ve süt verimi üzerinde etkili olan DGAT1 (diasilgliserol-asiltransferaz 1) geni üzerindeki polimorfik yapının Polimeraz Zincir Reaksiyonu (PZR) ve RFLP (Restriksiyon Enzimleri Uzunluk Polimorfizmi) yöntemleri ve PRNP (prion protein) geni üzerindeki polimorfik yapının Polimeraz Zincir Reaksiyonu (PZR) yöntemi kullanılarak ortaya konulması amaçlanmıĢtır.
Süt vücudumuzun ihtiyacı olan besin elementlerini en fazla bulunduran gıdadır. Bu besin elementleri organizma tarafından kolayca alınabilecek ve sindirilebilecek Ģekilde olup, organizmanın büyüme ve geliĢebilmesi için gerekli olan organik ve inorganik maddelerden oluĢmaktadır. Bu nedenle süt, sağlıklı ve dengeli beslenmede temel gıda maddesi olarak kabul edilmektedir. Ayrıca süt, kalsiyum, fosfor ve riboflavin açısından tüketilmesi gereken, vücut için gerekli olan doymamıĢ yağ asitlerini bünyesinde bulunduran süt yağı ile içerdiği yüksek kalitedeki süt proteini, mineraller ve vitaminlerle eĢsiz bir gıda maddesi olarak kabul edilmektedir. Sütte bulunan maddelerden bir kısmı diğer gıdalarda da bulunmasına rağmen laktoz, süt yağı, kazein, laktoglobulin ve laktalbumin1 hayvanın süt bezlerinde sentezlenmekte ve yalnız sütte bulunmaktadır.
Ayrıca süt proteini, doğada bulunan fakat organizmada yapılamadığı için dıĢarıdan alınması gereken löysin, izolöysin, valin, metiyonin, fenilalanin, treonin, triptofan, lizin amino asitlerini bol miktarda içermektedir, bu amino asitler besinlerdeki protein kalitesinin değerlendirilmesinde standart kabul edildiğinden, süt proteini kaliteli protein olarak değerlendirilmektedir (Ünal ve Besler 2006).
Ġnsan yaĢamında sağlıklı ve dengeli beslenmede, çok değerli olan süt ürünleri büyük ölçüde sığırlardan elde edilmektedir. Bu bakımdan dünya ve Türkiye’de sığır yetiĢtiriciliği hayvancılığın önemli bir kolunu oluĢturmaktadır.
Süt bileĢenlerinin içerisinde süt yağının neredeyse tamamını trigliseridler oluĢturur. Trigliserid sentezi, DGAT1 geni tarafından kodlanan asil CoA
28 diasilgliserol asiltransferaz 1 (DGAT1) enzimi tarafından katalizlenir (Grisart ve ark 2002).
Yüksek yapılı ökaryotlarda triasilgliserol metabolizmasını içeren lipoprotein oluĢumu ve intestinal yağ absorbsiyonu gibi fizyolojik süreçlerde temel bir rol oynayan (Casas ve ark 1998) asilCoA: diasilgliserol-asiltransferaz enzimini kodlayan gen, DGAT1 genidir (Strzalkowska ve ark 2005). Sığırlarda DGAT1 geni sütteki yağ yüzdesi etkisi bakımından güçlü bir aday gen, bununla birlikte bu özellik bakımından 14. sığır kromozomu üzerinde bulunan QTL bölgesine bulunmasından dolayı pozisyonel bir aday gendir (Winter ve ark 2002).
Yapılan çalıĢmalarda (Grisart ve ark 2002, Grisart ve ark 2004, Winter ve ark 2002, Weller ve ark 2003), sığırlarda, DGAT1 geninin ekson 8’de 10 433 ve 10 434 pozisyonunda bulunan adenin-adenin (AA) nükleotidlerinin, guanin-sitozin (GC) nükleotidlerine değiĢimi sonucunda asilCoA: diasilgliserol-asiltransferaz enziminin 232. pozisyonunda bulunan Lizin (K) amino asidinin yerine Alanin (A) amino asidinin geçtiği bir mutasyon belirlenmiĢtir.
DGAT1 geninin 10 230-10 644 nükleotitleri arasında mutasyonlar bulunmaktadır. Ancak bu mutasyonlar PZR tekniği ile belirlenemediğinden, sunulan çalıĢmada BI, YK, GAK, DAK ve ZAV ırklarının 10 433-10434 pozisyonundaki mutasyonu belirlemek amacıyla restriksiyon enzimi kullanılmıĢtır.
Literatürde çalıĢmamıza konu olan diğer gen olan PRNP promotor 23 bç ve intron 1 12 bç indel polimorfizmi ile süt verimi arasındaki iliĢkiyi inceleyen sadece bir yayın olup, bu araĢtırmada PRNP promotor 23 indel polimorfizmi ins/ins genotipi ile sütteki yağ ve protein miktarı arasında önemli bir korelasyon olduğu bulunmuĢtur. Ayrıca PRNP intron 1 12 bç indel polimorfizmini ile süt karakterleri arasında bir korelasyon saptanmamıĢtır (Czarnik 2007b). Literatürde yerli sığır ırklarında PRNP promotor 23 bç ve intron 1 12 bç indel polimorfizmini inceleyen yayın bulunmamaktadır.
29 Sunulan çalıĢmada, BI, YK, GAK, DAK ve ZAV ırklarına ait PRNP geninin 47784-47883 (promotor) ve 49686-49777 (intron 1) nükleotidleri arasını çoğaltmak amacıyla kullanılan primerlerin bağlandığı bölgeler arasında sırasıyla 23 bç ve 12 bç insersiyon-delesyon (indel) polimorfizminin olduğu bölge bulunmuĢtur. Bu nedenle PZR sonuçlarına bakarak indel tespit edilebilmiĢtir. Bundan dolayı PZR tekniği kullanılmıĢtır.
4.1. Bazı Yerli Sığır Irklarında PRNP Geni
Süt verimi bakımından yerli sığır ırklarımız karĢılaĢtırılırsa: bir laktasyon döneminde en yüksek süt verimine ZAV (4000-5000 kg), daha sonra GAK (2000-2500 kg), en düĢük süt verimi ise YK (700-800 kg) sığır ırkında saptanmıĢtır. Ek olarak DAK (1000-1200 kg) ve BI (1000-1500 kg) sığır ırkları süt verimi bakımından birbirine paralel özellik göstermektedir.
Sütteki yağ miktarı bakımından yerli sığır ırklarımızda en yüksek değer YK sığır ırkında (% 5), daha sonra DAK, BI, ve ZAV sığır ırklarında (% 4), en düĢük ise GAK (% 3) sığır ırkında belirlenmiĢtir (UlubaĢ ve Günay 2004).
Czarnik ve ark (2007b), PRNP promotor 23 bç ins/ins genotipi ile sütteki yağ ve protein miktarı arasında önemli bir korelasyon olduğunu saptamıĢlardır. Fakat bu özellik bakımından PRNP intron 1 12 bç ins/ins genotipi arasında bir korelasyon olmadığını belirtmiĢlerdir.
Sunulan çalıĢmada ise Çizelge 3.6.’daki veriler incelendiğinde, sütteki yağ miktarı özelliği ile ilgili olan PRNP promotor 23 bç ins/ins genotipinin (Czarnik ve ark 2007b) en yüksek frekansı çalıĢılan sığır ırkları arasında en yüksek sütteki yağ miktarı özelliğine sahip ZAV sığır ırkında (UlubaĢ ve Günay 2004) gözlendiği görülmektedir. Daha sonra sırası ile BI, YK, GAK ve DAK (sırası ile 0.32 – 0.10 – 0.08 – 0.07) sığır ırkında gözlenmiĢtir.
Ayrıca sunulan çalıĢmada DGAT1 K232A KK genotipi ile aynı etkiyi gösteren promotor ins/ins genotipinin bulgularının çeliĢtiği görülmektedir. PRNP
30 promotor ins/ins genotipinin en yüksek frekansı ZAV sığır ırkında gözlenirken, DGAT1 K232A KK genotipinin en yüksek frekansı BI ve DAK (0.38) sığır ırkında gözlenmiĢtir. Daha sonra ins/ins genotipi sırası ile BI, YK, GAK ve DAK sığır ırklarında gözlenirken KK genotipi ise GAK (0.25), ZAV (0.25), YK (0.18) sığır ırkında gözlenmiĢtir.
Juling ve ark (2006), PRNP promotor 23 bç ve intron 1 12 bç indel polimorfizmi ile BSE arasındaki iliĢkiyi incelemiĢlerdir. Haplotip analizi ile 23 del/12 ins ve 23 ins/12 ins arasında önemli bir fark olmadığını fakat 23 del/12 del haplotipinin BSE ile ilgili olduğunu göstermiĢlerdir. Ek olarak 12 del/del genotipinin BSE ana risk faktörü olduğunu belirtmiĢlerdir.
Ayrıca Sander ve ark (2004), PRNP promotor 23 bç indel polimorfizmi ile BSE arasında önemli bir korelasyonun olduğunu göstermiĢlerdir.
Seabury ve ark (2004) ise BSE’li ve sağlıklı hayvan grupları arasında, PRNP promotor 23 bç indel polimorfizmi bakımından önemli bir fark olmadığını fakat PRNP intron 1 12 bç indel polimorfizmi bakımından önemli bir fark olduğunu rapor etmiĢlerdir.
Sunulan çalıĢmada ise Çizelge 3.5’deki veriler incelendiğinde GAK, YK, BI, DAK ve ZAV sığır ırklarında PRNP promotor intron 1 12 bç ins/ins genotipinin del/del genotipinden daha yüksek olduğu görülmektedir. Bu nedenle çalıĢılan yerli sığır ırklarımız BSE riski taĢımamaktadır.
ÇalıĢılan yerli sığır ırklarımızda 23 del/12 del genotipi; GAK sığır ırkında 3, YK sığır ırkında 3, BI sığır ırkında 1 sığırda gözlenmiĢtir. Bu sığırlar yüksek BSE riski taĢımaktadır. DAK ve ZAV sığır ırkında söz konusu genotip gözlenmemiĢtir.
Süt verimi ve sütteki yağ miktarı üzerine etkisi olan DGAT1 genindeki ve sütteki protein ve yağ miktarı üzerine etkisi olan PRNP genindeki polimorfik yapıların sayısal olarak daha büyük populasyonda çalıĢılması ve aynı hedefe
31 yönelmiĢ farklı polimorfik yapıların sinerjik etkilerini ortaya koyan genetik faktörlerin belirlenmesi ıslah çalıĢmalarında önem arz etmektedir.
4.2. Bazı Yerli Sığır Irklarında DGAT1 Geni
GAK, YK, BI, DAK, ZAV sığır ırklarına ait DGAT1 K232A genotipik frekansı Hardy-weinberg dengesinden bir sapma göstermemiĢtir. Yani bu lokus bakımından allelik ve genotipik frekanslar arasında önemli bir fark yoktur. Sunulan çalıĢmada, çalıĢılan sığır ırkları arasında en düĢük süt verimi özelliğine sahip olan YK sığır ırkında, A allelin en yüksek değeri (0.58) gözlenmiĢtir. Bu değer çalıĢılan sığır ırkları arasında en yüksek süt verimine sahip olan Zavot ve GAK sığır ırklarına ait A allel frekansından (sırası ile 0.50 – 0.50) daha yüksek çıkmıĢtır. En düĢük A allel frekansı ise süt verimi birbirine yakın olduğu bilinen BI ve DAK sığır ırklarında (sırası ile 0.38 - 0.38) gözlenmiĢtir.
GAK, YK, BI ve DAK sığır ırklarında DGAT1 K232A polimorfizmini inceleyen Kerpek (2007)’in çalıĢmasının sunulan çalıĢmanın sonucu ile çeliĢtiği görülmektedir. Sunulan çalıĢmanın sonucu ile Kerpek (2007)’in sonucu arasında önemli bir farklılık bulunmaktadır. Sunulan çalıĢmada, çalıĢılan sığır ırkları arasında A allelin en yüksek frekansı YK sığır ırkında gözlenirken, Kerpek (2007)’in çalıĢmasında A allelin en düĢük frekansı YK sığır ırkında gözlenmiĢtir. Ayrıca sunulan çalıĢmada, çalıĢılan sığır ırkları arasında A allelin en düĢük frekansı DAK sığır ırkında gözlenirken, Kerpek (2007)’in çalıĢmasında A allelin en yüksek frekansı DAK sığır ırkında gözlenmiĢtir. Ek olarak sunulan çalıĢmada AA genotipi GAK sığır ırkında 7, YK sığır ırkında 8, BI sığır ırkında 4, DAK sığır ırkında 5, Zavot sığır ırkında 4 sığırda gözlenirken, Kerpek (2007)’in çalıĢmasında AA genotipi hiçbir ırkta gözlenmemiĢtir. Sonuçlara arasındaki farklılık, populasyondan populasyona farklılık olabileceğini göstermektedir. Kerpek (2007)’in bulguları da sunulan çalıĢmada GAK sığır ırkına ait A allel frekansının, BI sığır ırkına ait A allel frekansından daha yüksek olduğu sonucunu desteklemektedir.
Sonuç olarak: BI, YK, DAK, GAK ve ZAVOT ırklarında yürütülen bu çalıĢma ile elde edilen verilerin, bu sığır ırklarında süt özelliği ile DGAT1 ve PRNP
32 gen polimorfizmi arasındaki korelasyon ile ilgili çalıĢmalarda yararlı olacağı, hayvan yetiĢtiriciliği ve ıslah çalıĢmalarında süt verimi yönünden seleksiyona katkı sağlayacağı kanısına varılmıĢtır
33
5. SONUÇ ve ÖNERĠLER
Sonuç olarak; Türkiye’de bulunan bazı yerli sığır ırklarının DGAT1 K232A polimorfizmi PZR ve RFLP tekniği kullanılarak ve PRNP promotor 23 bç ve intron 1 12 bç indel polimorfizmi PZR tekniği kullanılarak belirlenmiĢtir. Yapılan istatistiksel analizlerle AK (heterozigot) genotipi bakımından sığır ırklarının kendi aralarında karĢılaĢtırılması sonucunda, grupların birbirine yakın dağılım gösterdiği ve istatistiksel bir farkın olmadığı, ins/del genotipleri bakımından ise istatistiksel bir farkın olduğu tespit edilmiĢtir.
DGAT1 geninde A allelin en yüksek frekansı YK (0,58) daha sonra GAK ve ZAV’da (0,50), en düĢük frekansı BI ve DAK’da (0,38) gözlenmiĢtir.
PRNP promotor ve intron 1 indel polimorfizmi için yapılan genotiplemede; promotor bölgesi en yüksek del/del genotipi frekansı DAK ve GAK (sırası ile 0,53-0,52), en düĢük BI ve ZAV (sırası ile 0,19 ve % 0,17) ırklarında gözlenmiĢ, Ġntron 1 bölgesi del/del genotipi en yüksek frekankansı ise BI ve GAK (sırası ile 0,25-0,13), daha sonra YK (0,08) en düĢük frekans DAK ve ZAV (0,05)’da gözlenmiĢtir.
BI, YK, DAK, GAK ve ZAVOT ırklarında yürütülen bu çalıĢma ile elde edilen verilerin, bu sığır ırklarında süt özelliği ile DGAT1 ve PRNP gen polimorfizmi arasındaki korelasyon ile ilgili çalıĢmalarda yararlı olacağı, hayvan yetiĢtiriciliği ve ıslah çalıĢmalarında süt verimi yönünden seleksiyona katkı sağlayacağı kanısına varılmıĢtır.
34
6.ÖZET
T.C.
SELÇUK ÜNĠVERSĠTESĠ SAĞLIK BĠLĠMLERĠ ENSTĠTÜSÜ
Türkiye’de Bulunan bazı sığır ırklarının DGAT1 ve PRNP Gen Polimorfizminin AraĢtırılması
Ġclal ġAHĠN
Biyokimya (VET) Anabilim Dalı
YÜKSEK LĠSANS TEZĠ / KONYA-2010
Sunulan çalıĢmada süt yağı ve süt verimi üzerine etkili olan DGAT1 geni K232A ve süt bileĢimine etki ettiği düĢünülen PRNP geninin promotor bölgesi (23 bç indel) ile intron 1 bölgesi (12 bç indel) insersiyon-delesyon (indel) polimorfizmi incelenmiĢtir.
Süt yağının neredeyse tamamını oluĢturan trigliserid sentezini katalizleyen DGAT1 enzimi kodlayan DGAT1 geni süt verim özelliği bakımından güçlü bir aday gen olarak belirlenmiĢtir.
ÇalıĢmada materyal olarak, Boz Irk (BI), Yerli Kara (YK), Doğu Anadolu Kırmızısı (DAK), Güney Doğu Anadolu Kırmızısı (GAK), ve Zavot ırklarından olmak üzere toplam 122 baĢ sığır kullanılmıĢtır.
Örnekler üzerinde DGAT1 geninin K232A polimorfizmi (K ve A alleller) PZR-RFLP tekniği kullanılarak ve PRNP geninin promotor bölgesi (23 bç indel) ile intron 1 bölgesi (12 bç indel) insersiyon-delesyon (indel) polimorfizmi PZR tekniği kullanılarak belirlenmiĢtir.
DGAT1 geninde A allelin en yüksek frekansı YK (0,58) daha sonra GAK ve ZAV’da (0,50), en düĢük frekansı BI ve DAK’da (0,38) gözlenmiĢtir.
PRNP promotor ve intron 1 indel polimorfizmi için yapılan genotiplemede; promotor bölgesi en yüksek del/del genotipi frekansı DAK ve GAK (sırası ile 0,53-0,52), en düĢük BI ve ZAV (sırası ile 0,19 ve 0,17) ırklarında gözlenmiĢ, Ġntron 1 bölgesi del/del genotipi en yüksek frekansı ise BI ve GAK (sırası ile 0.25-0,13), daha sonra YK (0,08) en düĢük frekans DAK ve ZAV (0,05)’da gözlenmiĢtir.
BI, YK, DAK, GAK ve ZAVOT ırklarında yürütülen bu çalıĢma ile elde edilen verilerin, bu sığır ırklarında süt özelliği ile DGAT1 ve PRNP gen polimorfizmi arasındaki korelasyon ile ilgili çalıĢmalarda yararlı olacağı, hayvan yetiĢtiriciliği ve ıslah çalıĢmalarında süt verimi yönünden seleksiyona katkı sağlayacağı kanısına varılmıĢtır.
35
7. SUMMARY
Investigation of DGAT1 and PRNP Genes Polymorphism of Various Cattle Breeds in Turkey
In the present study, K232A polymorphism of Diacyleglycerol-acyltransferase 1 (DGAT1) gene affecting milk production traits and insertion-deletion (indel) polymorphism with in the promoter sequence (23 bp indel) and intron 1 (12 bp indel) of the prion protein (PRNP) gene were studied.
DGAT1 gene coding DGAT1 enzyme that catalyzes the synthesis of triglycerides that forms almost all of milk fat was determined as a candidate gene with a strong effect on milk production traits. Samples of 122 animals from Turkish Grey (BI), Anatolian Black (YK), South Anatolian Red (GAK), East Anatolian Red (DAK) ve Zavot breeds were used for the study.
The samples were genotyped for DGAT1 K232A polymorphism (A and K allel) using the PCR-RFLP technique and PRNP gene polymorphism in the promoter region (23 bp indel) and intron 1 region (12 bp indel) using PCR analysis.
The highest frequency of A allele in DGAT1 gene was found in the YK (0,58) followed by GAK and ZAV (0.50), BI and DAK showed low frequencies of A alleles (0,38).
Genotypes for the promoter region and intron 1 of PRNP gene; in promoter region, the highest frequency of del/del genotype was found in the DAK and GAK (0.53 and 0,52 respectively) followed by YK (0.46), BI and ZAV showed low frequencies of del/del genotype (0,19 and 0.17 respectively). In intron 1 region the highest frequency of del/del genotype was found in the BI and GAK (0,25 and 0.13 respectively), followed by YK (0,08), DAK and ZAV (0.05) showed low frequencies of del/del genotype.
Results of the present study on BI, YK, DAK, GAK an ZAV breeds could be used to guide association studies between DGAT1 and PRNP genes polymorphism with milk traits in these breeds and may be useful selection for milk traits on animal husbandry and reclamation studies.
36
8. KAYNAKLAR
1. Batu S. Türkiye Sığır Irkları ve Sığır YetiĢtirme Bilgisi. Ankara, A.Ü.Vet.Fak.Yay. 1962,41. 2. Blott S, Kim JJ, Moisio S, Schmidt-Küntzel A. Molecular dissection of a quantitative trait locus. A
phenylalanine-to-tyrosine substitution in the transmembrane domain of the bovine growth hormone receptor is associated with a major effect on milk yield and composition. Genetics, 2003;163:253-266.
3. Campbell TA, Palmer MS, Will RG, Gibb WRG, Luthert PJ. A prion disease with a novel 96-base pair insertional mutation in the prion protein gene. Neurology, 1996;46 (3):761-766.
4. Casas E, White SN, Riley DG, Smith TPL, Brenneman RA, Olson TA, Johnson DD, Coleman SW, Bennett GL, Chase CC. Assessment of single nucleotide polymorphisms in genes residing on chromosomes 14 and 29 for association with carcass composition traits in Bos indicus cattle. J Anim Sci. 2005;83:13-19.
5. Cases S, Smith SJ, Zheng YW, Myers HM. Identification of a gene encoding an acyl CoA: diacylglycerol acyltransferase, a key enzyme in triacylglycerol synthesis. Proc Natl Acad Sci. 1998;95: 13018-13023.
6. Czarnik U, Zabolewicz T, Strychalski J, Grzybowski G, Bogusz M. Deletion-insertion polymorphism in the prion protein gene (PRNP) in Polish Holstein-Friesian cattle. J Appl Genet. 2007b;48:69-71.
7. Czarnık U, Strychalskı J, Bojarojć-Nosowıcz B, Kaczmarczyk E. Prıon proteın gene (PRNP) polymorphısm and ındıcators of secretıon dısorders of the mammary gland ın cows naturally ınfected wıth Bovıne Leukaemıa Vırus (Blv). Bull Vet Inst Pulawy. 2007a;51:459-464. 8. Czarnik U, Grzybowski G, Zabolewicz T, Strychalski J, Kamiński S. Deletion/insertion
polymorphism of the prion protein gene (PRNP) in Polish Red Cattle, Polish Whitebacked Cattle and European Bison (Bison bonasus L. 1758). Russian Journal of Genetics, 2009;45:453-459.
9. Ebi.ac.uk. DGAT1 geni nükleotid dizisi. EriĢim: http://www.ebi.ac.uk/cgi-bin/expasyfetch?AJ 31 8490 EriĢim tarihi; 11.10.2009.
10. Goldmann W, Hunter N, Martin T, Dawson H, Hope J. Different forms of the bovine PrP gene have five or six copies of a short G-C element within the protein coding region. Gen Virol. 1991;72:201-204.
11. Grisart B, Coppieters W, Farnir F, Karim L, Ford C, Berzi P, Cambisano N, Mni M, Reid S, Simon P, Spelman R, Georges M, Snell R. Positional candidate cloning of a QTL in dairy cattle: Identification of a missense mutation in the bovine DGAT1 gene with major effect on milk yield and composition. Genome Res. 2002;12:222-231.
12. Grisart B, Farnir F, Karim L, Cambisano N, Kim JJ, Kvasz A, Mni M, Simon P, Frere JM, Coppieters W. Genetic and functional confirmation of the causality of the DGAT1 K232A quantitative trait nucleotide in affecting milk yield and composition. Proc Natl Acad Sci. 2004;101:2398-2403.
13. Haase B, Doherr MG, Seuberlich T, Drögemüller C, Dolf G, Nicken P, Schiebel K, Ziegler U, Groschup MH, Zurbriggen A, Leeb T. PRNP promoter polymorphisms are associated with BSE susceptibility in Swiss and German cattle. BMC Genet. 2007;8:15.