TC.
EGE ÜNİVERSİTESİ TIP FAKÜLTESİ TIBBİ MİKROBİYOLOJİ ANABİLİM DALI
HCV GENOTİPİNİN BELİRLENMESİNDE 5’NCR VE NS5B BÖLGELERİNİN YENİ NESİL DİZİLEME (YND-NGS) İLE
ARAŞTIRILMASI
UZMANLIK TEZİ
Dr. Seniha AKTAKKA
DANIŞMAN:
Prof. Dr. Selda Erensoy
TC.
EGE ÜNİVERSİTESİ TIP FAKÜLTESİ TIBBİ MİKROBİYOLOJİ ANABİLİM DALI
HCV GENOTİPİNİN BELİRLENMESİNDE 5’NCR VE NS5B BÖLGELERİNİN YENİ NESİL DİZİLEME (YND-NGS) İLE
ARAŞTIRILMASI
UZMANLIK TEZİ
Dr. Seniha AKTAKKA
DANIŞMAN:
Prof. Dr. Selda Erensoy
DEĞERLENDİRME KURULU ÜYELERİ
(Adı Soyadı) (İmza)
Başkan : Prof.Dr. ………. ... (Danışman) Üye : Prof.Dr. ………. ... Üye : Prof.Dr. ………. ... Üye : Prof.Dr. ………. ... Üye : Prof.Dr. ………. ...
ÖNSÖZ
Ege Üniversitesi’ne başladığım ilk günde beni güler yüz ve sıcak kanlılıkla karşılayan başta Tıbbi Mikrobiyoloji Anabilim Dalı Başkanımız sayın Prof Dr.
Rüçhan Sertöz ve tüm öğretim üyesi hocalarıma emeklerinden dolayı teşekkür ederim.
Tezimin yazılması aşamasında bıkmadan büyük emek ve sabırla çalışan, sorduğum her soruya usanmadan cevap veren ve tecrübelerinden çok istifade ettiğim sayın Prof. Dr. Selda Erensoy hocama teşekkür ederim. Eğitimimde büyük katkıları olan Prof. Dr. Halil İbrahim Atabay ve Uzm. Dr.
Arzu Duran hocalarıma teşekkür ederim.
Uzmanlık eğitimimde beraber çalıştığımız, dostluk ve yardımlarını esirgemeyen Tıbbi Mikrobiyoloji asistanı arkadaşlarıma, laboratuvarda birlikte çalıştığımız laboratuvar teknisyenleri ve personele, özellikle tez çalışmamda büyük desteği olan laboratuvar teknisyeni Yaşar Gökçe’ye
teşekkür ederim.
Öğrenciliğim ve asistanlığım sırasında her zaman desteklerini yanımda hissettiğim, her konuda olduğu gibi okumamda da üzerimde çok büyük
emekleri olan annem ve babama, varlıklarıyla her zaman mutluluk duyduğum, hayatımın çok önemli birer parçası olan kardeşlerime teşekkür
ederim.
En önemlisi, hem evde hem hastanede hayatımın her aşamasında yanımda olan, desteğini her zaman arkamda hissettiğim ve bana her konuda çok yardımcı olan sevgili eşim Yunus’a ve gelmesini heyecanla ve sabırsızlıkla beklediğimiz, şimdiden hayatımıza renk ve mutluluk katan oğlumuz Eymen’e
çok teşekkür ederim.
I ÖZET
Giriş ve Amaç: Hepatit C virüsü (HCV) çok yüksek derecede genetik çeşitliliğe sahiptir. HCV suşları, viral genomların filogenetik ve sekans analizleri temelinde yedi tanınmış genotipe ayrılır. HCV’de genotip belirlenmesi hastaya uygun tedavinin seçilmesinde önemlidir. Özellikle doğrudan etkili antivirallerin tedavide kullanıma girmesiyle genotiplerin ve alt tiplerin doğru belirlenmesi daha da önemli hale gelmiştir. HCV genotiplemesi için altın standart tam genomun nükleik asit dizi analizidir. Pratikte 5’NCR, kor ve NS5B gen bölgeleri genotiplemede analiz edilmektedir. Nükleik asit dizi analizi için günümüzde en sık kullanılan yöntem olan Sanger Dizi Analiz Yöntemi zincir sonlandırma reaksiyonu olarak bilinir. Yeni nesil dizileme (YND) sistemleri ile ise bir çalışmada tüm genom, transkriptom veya daha küçük hedef bölgelerdeki milyonlarca DNA parçası dizilenebilmektedir. YND teknolojisinin Sanger dizileme yöntemine göre daha hızlı ve yüksek verimli sonuçlar verdiği görülmektedir. Ege Üniversitesi Tıp Fakültesi Tıbbi Mikrobiyoloji Anabilim Dalı Moleküler Viroloji Laboratuvarı’nda 2000 yılından beri HCV genotip testi farklı yöntemlerle rutin olarak yapılmaktadır. Laboratuvarımızda rutinde son olarak genotipe özgü gerçek zamanlı PCR ve/veya NS5B ve/veya 5’NCR bölgelerinin Sanger zincir sonlandırma reaksiyonu dizilemesi ile HCV genotipi belirlenmektedir. Bu çalışmada daha hızlı ve yüksek verimli olan YND ile HCV 5’NCR ve NS5B bölgeleri kullanılarak genotipleme yapılması ve laboratuvarımıza uygun HCV genotipleme yönteminin belirlenmesi amaçlanmaktadır.
Gereç ve yöntem: Laboratuvarımıza HCV genotip testi için gönderilmiş ve -80˚C’de arşivlenmiş olan 60 HCV pozitif hastanın plazma örneği çalışmamıza dahil edilmiştir. RNA ekstraksiyonu, tersine (revers) transkripsiyon basamaklarından sonra, 5’NCR bölgesi ve NS5B bölgesi için ayrı ayrı dış PCR, nested (ikinci tur) PCR ve analiz basamakları
II uygulanmıştır. HCV genomunun 5’NCR ve NS5B bölgelerinin PCR ürünlerinin ABI 3500 Genetic Analyzer (Applied Biosystems Foster City, CA, ABD) cihazı ve MiSeq (Illumina, San Diego, CA, ABD) (YND) ile analizi yapılmış, nükleik asit dizileri NCBI gen bankasındaki genotipleme bölümü (https://www.ncbi.nlm.nih.gov/projects/genotyping/formpage.cgi)
kullanılarak genotipleme yapılmıştır.
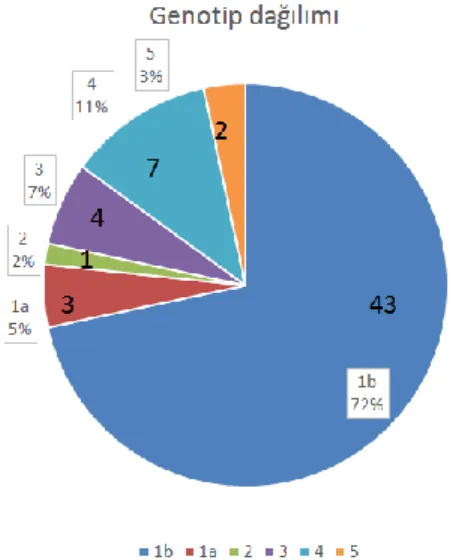
Bulgular: Ellibeş örnekte NS5B bölgesinin hem YND ve Sanger ile, hem de 5’NCR bölgesinin YND ve Sanger ile genotip sonuçları benzer bulundu. (%91,67). YND ile elde edilen verilerin analizine göre; NS5B bölgesi dizi analizi ile 60 örneğin 43’ü (%71,67) genotip 1b, yedisi (%11,67) genotip 4, dördü (%6,67) genotip 3, üçü (%5) genotip 1a, ikisi (%3,33) genotip 5, biri (%1,67) genotip 2 olarak saptandı. 5’NCR bölgesi dizi analizi ile ise, 60 örneğin 36’sı (%60) genotip 1b, yedisi (%11,67) genotip 4, dördü (%6,67) genotip 3, beşi (%8,33) genotip 1a, ikisi (%3,33) genotip 5, ikisi (%3,33) genotip 6, biri (%1,67) genotip 2 olarak saptandı. 5’NCR bölgesi dizi analizi ile üç örnek (%5) alt tip ayrımı yapılamadan genotip 1 olarak belirlendi.
Her iki bölgenin analizine göre en çok saptanan genotip, genotip 1b oldu. Çalışmada genotip 7 olarak tanımlanan örnek olmadı.
Sanger dizi analizi ve YND ile elde edilen diziler örnek bazında hizalandığında; YND ile 38-40 baz daha uzun temiz dizi elde edildiği görüldü. 5’NCR bölgesine ait dizilerde 60 örnekten 53’ünün (%88,3) %100 uyumlu, beşinin (%8,3) %99 uyumlu, birinin (%1,6) %98 uyumlu, birinin (%1,6) %97 uyumlu olduğu görüldü. NS5B bölgesine ait dizilerde ise 60 örnekten; 45’i (%73,3) %100 uyumlu, beşi (%8,3) %99 uyumlu, biri (%1,6) %98 uyumlu, dördü (%6,6) %95-97 uyumlu, dördü (%6,6) %92-94 uyumlu, biri (%1,6) %83 uyumlu idi.
III Sanger zincir sonlandırma reaksiyonu ve YND ile elde edilen verilerden her iki grupta da genotip 1 olarak tanımlanan üç örnekte 5’NCR bölgesi dizi analizi ile alt tip ayrımı yapılamazken, bu örnekler NS5B bölgesi dizi analizi ile genotip 1b olarak saptandı. İki örnek, iki yöntemle de 5’NCR bölgesi dizi analizinde genotip 1a, NS5B bölgesi dizi analizinde genotip 1b; iki örnek 5’NCR bölgesi dizi analizi ile genotip 6, NS5B bölgesi dizi analizi ile genotip 1b olarak saptandı. Bu durum; bilindiği gibi genotip 1 alt tiplerinin belirlenmesinde ve 1 ile 6 ayrımında NS5B bölgesinin daha güvenilir olmasına bağlandı.
Sanger YND dizilerinin hizalamasında NS5B dizisi %83 uyumlu olan örnek Sanger ile yapılan NS5B bölgesi dizi analizinde genotip 1b, Sanger zincir sonlandırma reaksiyonu 5’NCR, YND NS5B ve YND 5’NCR bölgeleri dizi analizlerinde genotip 1a olarak saptandı. Bu durum Sanger ile elde edilen NS5B dizisinin temiz okunan kısmının kısa olmasına bağlandı.
Sonuç: Altmış örneğin 59’unda hem NS5B, hem de 5’NCR dizileri YND ve Sanger analizleriyle aynı sonuçları vermiştir (%98,33 uyum). YND ile örneklerin havuzlanması ve çoklu çalışılması (“multipleks”) ile maliyet düşmektedir. Klinik açıdan önemli olan genotip belirlenmesi, NS5B bölgesinin YND gibi güvenilir bir yöntemle analiziyle etkin olarak yapılabilmektedir. Değerlendirmelerin sonucuna göre; 60 örneğin 43’ü genotip 1b (%71,67), üçü 1a (%5), yedisi 4 (%11,67), dördü 3 (%6,67), ikisi 5 (%3,33), biri 2 (%1,67) bulunmuştur.
IV ABSTRACT
Introduction and AIM: Hepatitis C virus (HCV) has very high genetic diversity. HCV strains are divided into seven known genotypes based on phylogenetic and sequence analysis of viral genomes. Genotype identification of HCV is important in determining the appropriate treatment of the patient and the duration of the treatment. In particular, direct identification of genotypes and subtypes have become even more important as direct-acting antivirals become available for treatment. Nucleic acid sequence analysis of the whole genome is the gold standard for HCV genotyping. In practice, NS5B, core, and 5’NCR regions are analysed for genotyping. HCV genotype test has been routinely performed since 2000 in the Molecular Virology Laboratory of the Department of Medical Microbiology, Faculty of Medicine, Ege University. HCV genotyping with Sanger chain termination reaction is currently routinely being performed from HCV positive patient samples. In this study it is aimed to evaluate the eficiency of genotyping by using HCV 5’NCR and NS5B regions with next generation sequencing (NGS) and to determine the appropriate HCV genotyping method in our laboratory.
Materials and methods: Sixty archived (at -80°C) HCV positive plasma samples which were sent for HCV genotyping to the laboratory were included in the study. RNA extraction, reverse transcription steps followed by external PCR, nested (second round) PCR and analysis steps for the 5’NCR region and NS5B region separately. PCR products of the 5’NCR and NS5B regions of the HCV genome were analyzed with the ABI 3500 Genetic Analyzer (Applied Biosystems Foster City, CA, USA) and the MiSeq (Illumina, San Diego, CA, USA) (NGS), and the nucleic acid sequences were genotyped at the NCBI gene bank (https://www.ncbi.nlm.nih.gov/projects/
V Results: According to the analysis of data obtained with NGS; by NS5B region sequence analysis 43 (72%) of the 60 samples were genotype 1b, seven (11%) genotype 4, four (7%) genotype 3, three (5%) genotype 1a, two (3%) genotype 5, one (2%) was genotype 2. With the 5’NCR region sequence analysis; 36 (60%) of the 60 samples were genotype 1b, seven (12%) genotype 4, four (7%) genotype 3, five (8%) genotype 1a, two (3%) genotype 5, two (3%) genotype 6, one (2%) was genotype 2. With 5’NCR region sequence analysis, three samples (5%) were identified as genotype 1 without subtype discrimination. According to analysis of both regions, genotype 1b was the most detected genotype. There was no sample defined as genotype 7.
With NGS, 38-40 base longer clean sequences could be obtained compared to the Sanger sequences. When Sanger and NGS sequences were aligned on sample basis; among the 5'NCR region sequences, 53 (88,3%) of the 60 samples had 100% identity, five of them (8,3%) had 99% identity, one of them (1,6%) had 98% identity, one of them (1.6%) had 97% identity. Among the NS5B region sequences, 45 (73.3%) of the 60 samples had 100% identity, five (8.3%) had 99% identity, one (1.6%) had 98% identity, four (6.6%) had 95-97% identity, 4 (6.6%) had 92-94% identity, one (1.6%) had 83% identity. The 83% identical sample was determined as genotype 1a with Sanger and as genotype 1b with NGS. This sample was identified as genotype 1a by 5 'NCR sequence analysis both with NGS and Sanger.
Sanger chain termination reaction and NGS data in both groups showed that; three samples identified as genotype 1, while subtype discrimination could not be done with 5’NCR region sequence analysis, these samples were identified as genotype 1b with NS5B region sequence analysis. Two samples were identified as genotype 1a with 5’NCR region sequence analysis, but identified as genotype 1b with NS5B region sequence analysis.
VI Two samples were identified as genotype 6 with 5’NCR region sequence analysis but identified as genotype 1b with NS5B region sequence analysis. It was thought to be associated to the insufficiency of 5’NCR seuence for subtype determination and discrimination of genotype 6 from some genotype 1 strains as it has been known.
One sample was identified as genotype 1b by NS5B region sequence analysis with the Sanger chain termination reaction, but identified as genotype 1a by 5’NCR region sequence analysis with Sanger, NS5B region sequence analysis and 5’NCR region sequence analysis with NGS. The NS5B sequence which could be obtained by Sanger method was short; it was thought to be the reason of incompatibility.
Conclusion: Next generation sequencing and Sanger chain termination results were the same both with NS5B and 5’NCR regions in 59 of 60 samples (98.33). Cost was reduced with NGS by pooling and "multiplexing" the samples. Analysing NS5B region with a reliable method such as NGS is efficient in determining the genotypes which is clinically important to guide the treatment. According to overall evaluation of the data; 43 of 60 samples (71.67%) were genotyped as genotype 1b, three were genotype 1a (5%), seven were genotype 4 (11.67%), four were genotype 3 (6.67%), two were genotype 5 (3.33%), an done was genotype 2 (1.67%).
VII İÇİNDEKİLER ÖZET……… ... ... I ABSTRACT ... ... III İÇİNDEKİLER ... ... V ŞEKİLLER…… ... ... VII TABLOLAR……… ... ... VIII KISALTMALAR……… . ... IX 1. GİRİŞ ve AMAÇ……… ... 1 2. GENEL BİLGİLER ……… ... 2 2.1.Hepatit C virüsü……… ... 2 2.1.1.Tarihçe ……… . 2 2.1.2.Sınıflandırma ... 3
2.1.3.Hepatit C Virüsünün Genel Özellikleri ... 4
2.1.3.1.HCV’nin Genomik Yapısı ... 5
2.1.3.1.1.HCV’nin "Translasyon Olmayan" Bölgeleri 7 2.1.3.1.2.HCV’nin "Protein Kodlayan" Bölgesi ... 7
2.1.3.2.HCV’nin Yaşam Döngüsü… ... 11
2.1.1.1.1. HCV Replikasyonu……… ... 12
2.1.4.HCV Epidemiyolojisi ve Bulaş Yolları ... 13
2.1.5.HCV Enfeksiyonunda Patogenez Ve İmmun Yanıt... 14
2.1.6.Viral Genotipler ... 15
2.1.7.HCV Enfeksiyonunun Kliniği ……… ... 17
2.1.7.1.Akut Hepatit C ... 18
2.1.7.2.Kronik Hepatit C ... 19
2.1.8.HCV Enfeksiyonunda Özgül Mikrobiyolojik Tanı ... 19
2.1.8.1.Serolojik Yöntemler ... 19
2.1.8.2.Moleküler Yöntemler ... 21
VIII
2.1.8.3.1.Sanger Zincir Sonlandırma Reaksiyonu .... 26
2.1.8.3.2.Yeni Nesil Dizileme………... 28
2.1.9.HCV Enfeksiyonunda Tedavi……… ... 31
3. GEREÇ VE YÖNTEM ... 35
3.1.RNA Ekstraksiyonu ... 36
3.2.Revers Transkripsiyon ... 37
3.3.Polimeraz Zincir Reaksiyonları ... 37
3.3.1.5’NCR Bölgesi İçin Uygulanan PCR ... 37
3.3.2.NS5B Bölgesi İçin Uygulanan PCR ... 38
3.3.3.Sekans PCR ... 40
3.3.4.Saflaştırma ……… ... 41
3.4.Yeni Nesil Dizileme ... 41
3.4.1.PCR Ürünlerinin Pürifikasyon İşlemi ... 41
3.4.2.DNA Tegmentasyonu ve PCR Amplifikasyonu ... 42
3.4.3.PCR Temizleme ... 43
3.4.4.MiSeq (Illumina, San Diego, CA, ABD) Cihaza Yükleme 43 3.4.5.Veri Analizi ... 44 4. BULGULAR………..………..……… ... 45 5. TARTIŞMA………..…… ... 60 6. SONUÇ………. ... 72 7. KAYNAKLAR………..…………..……….… ... 73 8. ÖZGEÇMİŞ………..………..………. ... 87 9. EKLER………..………..……….… ... 88
IX ŞEKİLLER
Şekil 1. HCV viriyon yapısı ... 4
Şekil 2. HCV genom yapısı ve kodlanan proteinler ... 5
Şekil 3. HCV yaşam döngüsü şematik diyagra ... 11
Şekil 4. 6 büyük HCV genotipinin coğrafik yaygınlığı ... 17
Şekil 5. Hepatit C virüsü enfeksiyonunun doğal seyri ... 18
Şekil 6. Birinci, ikinci ve üçüncü kuşak ELISA testlerinin araştırdığı antijenler ... 20
Şekil 7. Sanger dizileme………..…….……….. ... 27
Şekil 8. Illumina teknolojisinde DNA dizileme basamakları ... 30
Şekil 9. HCV üzerine direkt etkili antivirallerin etki mekanizması ... 34
Şekil 10. HCV tam genom üzerinde primerlerin bağlanması beklenen kısımlar ... 40
Şekil 11. YND NS5B dizi analizine göre genotip dağılımı ... 49
Şekil 12. NS5B bölgesinde Genotip 1 için ayırt ettirici bölgeler ... 52
Şekil 13. NS5B bölgesinde Genotip 2 için ayırt ettirici bölgeler ... 52
Şekil 14. NS5B bölgesinde Genotip 3 için ayırt ettirici bölgeler ... 53
Şekil 15. NS5B bölgesinde Genotip 4 için ayırt ettirici bölgeler ... 53
Şekil 16. NS5B bölgesinde Genotip 5 için ayırt ettirici bölgeler ... 54
Şekil 17. NS5B bölgesinde Genotip 6 için ayırt ettirici bölgeler ... 54
Şekil 18. YND 5’NCR dizilerine göre filogenetik ağaç ... 58
Şekil 19. YND NS5B dizilerine göre filogenetik ağaç ... 59
X TABLOLAR
Tablo 1. Onaylanmış HCV genotipleri / alt tipleri ... 3
Tablo 2. HCV genomunun kodladığı proteinler ve işlevleri ... 6
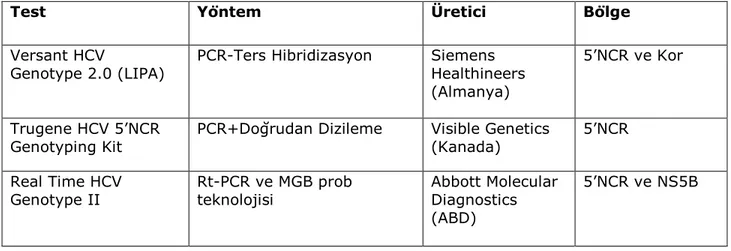
Tablo 3. Ticari genotipleme yöntemler ... 26
Tablo 4. Sanger ve YND ile belirlenen genotip sonuçları ... 46
Tablo 5. YND 5’NCR ve NS5B bölgeleri dizi analizine göre belirlenen genotip sayıları ... 50
Tablo 6A. 5’NCR YND ile elde edilen dizilerin evrimsel uzaklık ve benzerlik oranları ... 55
Tablo 6B. NS5B YND ile elde edilen dizilerin evrimsel uzaklık ve benzerlik oranları ... 56
XI KISALTMALAR
HCV: Hepatit C virüsü YND: Yeni Nesil Dizileme
NGS: Next generation sequencing 5’NCR: 5’Non-coding region
NS5B: Nonstructural protein 5B DEA: Doğrudan etkili antiviral IFN: İnterferon
AHC: Akut hepatit C KHC: Kronik Hepatit C
HCC: Hepatoselüler karsinom
HCV-RNA: Hepatit C Virüs Ribonükleik asit PCR: Polimeraz zincir reaksiyonu
dNTP: deoksinükleosittrifosfat ddNTP: Dideoksinükleosittrifosfat
RFLP: Restriction Fragment Length Polymorphism CDC: Centers for Disease Control and Prevention ICVT: Uluslarası Virüs Taksonomisi Birliği
ORF: Open Reading Frame
IRES: Internal ribosomal entry site LDL: Düşük yoğunluklu lipoprotein VLDL: Çok düşük yoğunluklu lipoprotein HVR: Hyper Variable Region
XII CTL: Sitotoksik T lenfosit
NS2: Nonstructural protein 2 NS3: Nonstructural protein 3 NS4A: Nonstructural protein 4A NS4B: Nonstructural protein 4B NS5A: Nonstructural protein 5A RdRp: RNA bağımlı RNA polimeraz SR-B1: Scavenger reseptor B tip 1 ALT: Alanin aminotransferaz
EIA: Enzyme immunoassay
ELISA: Enzyme-linked immunosorbent assay CIA: Chemiluminescence Immunoassay IU: International unit
RT-PCR: Gerçek zamanlı PCR
TMA: Transcription-Mediated Amplification cDNA: Komplementer DNA
bDNA: Dallı zincirli DNA MGB: Minor groove binder KVY: Kalıcı virolojik yanıt EVY: Erken virolojik yanıt
FDA: Food and Drug Administration (Amerikan Gıda ve İlaç Dairesi) PEG: Monometoksi polietilen glikol
RBV: Ribavirin
NPI: NS5B nükleozid inhibitörleri
XIII PI: Proteaz inhibitörleri
DS: Doğrudan sekanslama UDPS: Ultra-deep sequencing µl: Mikrolitre
NK: Natural Killer ∞: Sonsuz
1 1. GİRİŞ VE AMAÇ
Hepatit C virüsü (HCV) akut ve kronik hepatite neden olan kan yoluyla bulaşan bir virüstür (1). HCV, non-A, non-B hepatit virüsü enfeksiyonlarının çoğundan sorumludur. HCV enfeksiyonu vakaların %85’inde kronik enfeksiyona neden olan virüs kalıcılığı ile karakterizedir (2). Dünya çapında HCV’ye yakalanmış 185 milyondan fazla insanın bulunduğu tahmin edilmektedir (3). Yetmiş bir milyon bireyin ise kronik hepatit C hastası olduğu tahmin edilmektedir (4).
HCV, akut hepatitlerde %20, kronik hepatitlerde ise %70 etken olarak sorumlu tutulmaktadır. Akut ve kronik HCV enfeksiyonlarının genellikle asemptomatik ve subklinik seyretmesi nedeni ile tanısı her zaman konulamamaktadır. HCV enfeksiyonlarının %75-90’ı kronikleşmekte, bunların %20’sinde siroz gelişmekte, siroz gelişenlerde ise yıl başına ortalama %1-4 oranında hepatosellüler karsinom (HCC) gelişme riski bulunmaktadır (5).
HCV’nin başlıca bulaş yollarının, kan ve kan ürünleri ile direk perkütan temas ve damar içi uyuşturucu madde bağımlılarında ortak iğne kullanımı olduğu gösterilmiştir. HCV enfeksiyonu riskini arttıran diğer faktörler arasında; uzun süre hemodiyalize girme, büyük cerrahi girişim öyküsü, tek kullanımlık olmayan iğne kullanımı ve uzun süreli onkoloji ya da hematoloji kliniklerinde yatma yer almaktadır (6,7).
HCV çok yüksek derecede genetik çeşitliliğe sahiptir. HCV suşları, viral genomların filogenetik ve sekans analizleri temelinde yedi tanınmış genotipe ayrılır. Farklı genotiplere ait HCV türleri, nükleotid alanlarının %30-35’inde farklılık gösterir. HCV her bir genotip içinde, ayrıca onaylanmış 67 ve geçici 20 alt tipte sınıflandırılmıştır. Aynı alt türe ait suşlar, nükleotid alanlarının <%15’inde farklılık gösterir (8,9).
HCV genotiplerinden özellikle 1a, 1b, 2a ve 3a alt türlerinin yaygın olarak dünyaya dağılmış olduğu ve yüksek gelirli ülkelerde HCV enfeksiyonlarının büyük bir bölümünü oluşturduğu tespit edilmiştir (10).
2 Ülkemizde de dünyadaki verilerle benzer olarak en yaygın genotipin genotip 1b olduğu gösterilmiştir (11–20).
HCV’de genotip belirlenmesi, hastaya uygun tedavinin ve uygulanacak olan tedavinin süresinin belirlenmesinde ve bu tedaviye hastanın verdiği yanıtın izlenmesinde önemlidir. HCV enfeksiyonunda rutin olarak kullanılan tedavi Peg interferon + ribavirin ile kombine tedavidir. Bu tedaviye yanıtı belirleyen en önemli faktörler, hastanın enfekte olduğu HCV suşunun genotipi ve başlangıçtaki HCV-RNA viral yüküdür. HCV genotip 1 ile oluşan enfeksiyonlarda kalıcı viral yanıt %42-46 arasında, genotip 2 ve 3’de oranlar %76-82 arasında değişmektedir (17). HCV’ye doğrudan etkili antivirallerin tedavide kullanıma girmesiyle genotiplerin ve alt tiplerin doğru belirlenmesi daha da önemli olmuştur.
HCV genotiplemesi için altın standart nükleik asit dizi analizidir. Viral genomun tamamı kullanılarak yapılan tiplendirme ideal olmasına rağmen bu pratik bir yöntem olmadığı gibi maliyeti de yüksektir. Bu yüzden çoğunlukla, yeterli düzeyde farklılaşmış genom bölgesi polimeraz zincir reaksiyonu (PCR) ile çoğaltılarak dizi analizi yapılmaktadır (11).
Sanger Dizi Analiz Yöntemi zincir sonlandırma reaksiyonu olarak bilinir. Dideoksinükleosittrifosfatlar (ddNTP’ler) OH grubu taşımayan modifiye nükleotid substratlardır. Bunlar riboz şekerinin üçüncü karbon atomunda deoksi halde olduğundan fosfodiester bağının oluşamaz ve yeni nükleotidler yapıya katılamaz. DNA zincir uzaması sonlanır. Sonuçta primer sonundan başlayıp erken sonlanmaların olduğu bölgelere kadar çeşitli uzunlukta DNA parçaları oluşur. Bu parçalar birleştirilerek dizileme yapılır (21,22).
Yeni nesil dizileme (YND) sistemleri ile bir çalışmada tüm genom, transkriptom veya daha küçük hedef bölgelerdeki milyonlarca DNA fragmenti dizilenebilmektedir (23,24). YND teknolojisinin Sanger dizileme yöntemine göre daha hızlı ve yüksek verimli sonuçlar verdiği görülmektedir.
Ege Üniversitesi Tıp Fakültesi Tıbbi Mikrobiyoloji Anabilim Dalı Moleküler Viroloji Laboratuvarında 2000 yılından beri HCV genotipinin
3 belirlenmesi rutin olarak yapılmaktadır. Laboratuvarımızda RFLP analizi, PCR ürünlerinin Sanger zincir sonlandırma reaksiyonu ile dizi analizi ve genotipe özgü gerçek zamanlı PCR yöntemleri kullanılmıştır. Hepatit C tedavisinde doğrudan etkili antivirallerin kullanıma girmesi ile HCV genotiplerinin alt tiplerinin belirlenmesi büyük önem kazanmıştır. Genotip alt tiplerinin belirlenmesinde HCV genomunun farklı bölgelerinin analizinin etkinliği değişmektedir. Sanger yöntemiyle dizi analizinde kullanılmakta olan 5’NCR bölgesi özellikle genotip 1’in alt tiplerinin belirlenmesinde yetersiz kalmaktadır. Bu nedenle NS5B bölgesinin de analizine başlanmıştır.
Bu çalışmada daha hızlı ve yüksek verimli olan YND ile HCV 5’NCR ve NS5B bölgeleri kullanılarak genotipleme yapılması ve laboratuvarımıza uygun HCV genotipleme yönteminin belirlenmesi amaçlanmaktadır.
4 2. GENEL BİLGİLER
2.1. Hepatit C virüsü 2.1.1. Tarihçe
HCV, akut veya kronik karaciğer enfeksiyonuna neden olan tek hepatotropik flavivirüstür. 1970’li yıllardan beri Hepatit A ve Hepatit B virüslerinin dışında, başka virüslerin de akut veya kronik hepatite neden oldukları düşünülmüş ve bu olgular ne-A, ne-B hepatiti olarak adlandırılmıştır (25). HCV’nin varlığı 1988 yılında, CDC ve Chiron firması araştırmacılarının birlikte yaptıkları çalışmada, ne-A, ne-B hepatitli insan kanı ile kontamine edilmiş faktör VIII ile enfekte edilen şempanzelerin serum örneğinden klonlanarak gösterilmiştir (26). Viral genomun tam olarak açıklanmasından sonra HCV, genom yapısı ve replikasyon siklusu benzerlikleri nedeni ile,
Flaviviridae ailesi içinde, flavivirüs ve pestivirüslerden sonra üçüncü bir sınıf
olarak Hepacivirus genusunda sınıflandırılmıştır (27). 2.1.2. Sınıflandırma
Hepatit C virüsü, Flaviviridae ailesinin Hepacivirus genusu içerisinde yer almaktadır. VIII. Uluslarası Virüs Taksonomisi Birliği (ICVT)’nin 2005 yılında yayınladığı uzlaşı raporuna göre HCV GB virüs B ile birlikte
Flaviviridae ailesinin Hepacivirus cinsi içinde tanımlanmıştır (28). HCV
genomunun DNA veya protein dizilimi karşılaştırılarak yapılan çalışmalara göre 7 genotip ve bu genotipler içerisinde çok sayıda alt tipin (onaylanmış 67 ve geçici 20 alt tip) var olduğu ortaya konulmuştur (8,9). Genotipler Arap rakamları ile (1,2,3,..), alttipleri ise küçük Latin harfleri ile (a,b,c,..) gösterilmektedir (11). Tablo 1’de onaylanmış HCV genotipleri ve alt tipleri görülmektedir.
5 Tablo 1. Onaylanmış HCV genotipleri / alt tipleri
Hepatit C virüsünde diğer RNA virüslerinde olduğu gibi RNA bağımlı RNA polimeraz enziminin düzeltme aktivitesi bulunmamaktadır. Bu nedenle genom düzeyinde değişkenliği fazladır. Kronik HCV hastalarında, başta homojen olan virüs populasyonu, yeni mutant suşların çıkması ile heterojen duruma gelmekte ve bu mutant suşlar “quasispecies” (türümsü) olarak adlandırılmaktadır. HCV genom yapısının enfekte konakta oluşan bu özelliği sayesinde virüs, yaşadığı ortama çok iyi adaptasyon sağlamakta ve enfeksiyonun sürekliliği sağlanmaktadır (29).
2.1.3. Hepatit C Virüsünün Genel Özellikleri
Hepatit C virüsü küresel, lipid zarflı, yaklaşık 50 nm çapında, pozitif polariteli, tek iplikli bir RNA virüsüdür (25). HCV’nin immünelektron mikroskobu ile görüntülenmesi başarılmış, 55-65 nm çapında ve üzerinde zarfı delerek çıkan, ince dikensi yapılar taşıyan partiküller gösterilmiştir (25,30).
Kor proteini ve E1 ve E2 kılıf glikoproteinleri bir virionun ana protein komponentleridir. Genomik RNA’nın dışı birçok kor proteininin oluşturduğu
Genotip Alt tip
1 a, b, c, e, g, h, l 2 a, b, c, d, e, i, j, k, m, q, r 3 a, b, g, h, i, k 4 a, b, c, d, f, g, k, l, m, n, o, p, q, r, t, v, w 5 a 6 a, b, c, d, e, f, g, h, i, j, k, l, m, n, o, p, q, r, s, t, u, v, w, xa 7 a
6 nükleokapsit ve bunun dışında E1 ve E2 proteinlerinin bağlandığı konak hücrenin çift-katmanlı lipit tabakası ile kaplıdır (31). (Şekil 1)
Şekil 1. HCV viriyon yapısı (32)
Hepatit C virüsü infekte ettiği konakçıda değişik formlarda dolaşır. Düşük yoğunluklu lipoprotein (LDL) , çok düşük yoğunluklu lipoprotein (VLDL), veya immünoglobülinlere bağlı olabildiği gibi serbest formda da bulunabilir (33).
2.1.3.1. HCV’nin Genomik Yapısı
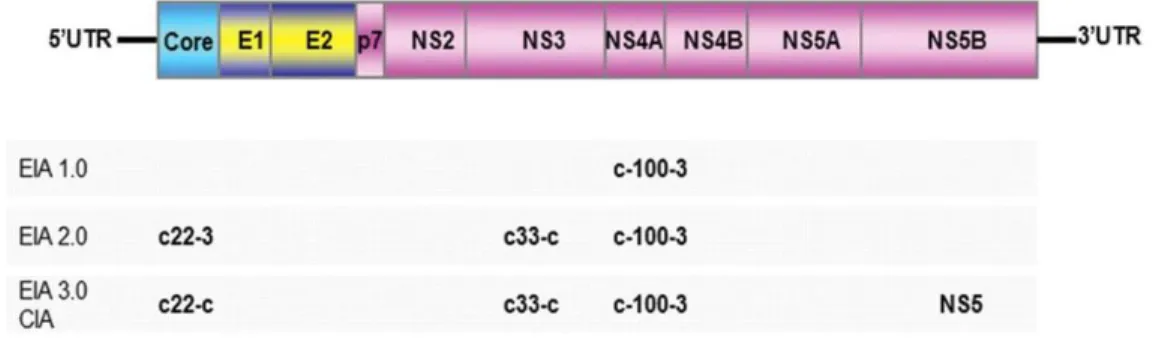
Hepatit C virüsü yaklaşık 9600 nükleotid uzunluğunda, pozitif polariteli, tek iplikli bir RNA taşımaktadır. Genom yapısını her iki uçta bulunan, herhangi bir protenini kodlamayan 5’ ve 3’NCR (non-coding region; NCR ya da untranslated region; UTR) bölgeleri ve bunların arasında bulunan ORF (Open Reading Frame, Açık Okuma Çerçevesi) bölgesi oluşturmaktadır. (Şekil 2) ORF bölgesi 330. nükleotiddeki ilk ATG’de başlar, 9377 nükleotidden oluşur ve hemen hemen tüm genomu kapsar. 3011 aminoasit uzunluğunda büyük bir poliprotein kodlar (25,28,34,35). Tablo 2’de HCV genomunun kodladığı proteinler ve işlevleri gösterilmiştir.
7 Şekil 2. HCV genom yapısı ve kodlanan proteinler (28)
Tablo 2. HCV genomunun kodladığı proteinler ve işlevleri
Protein Aminoasit Aralığı İşlevi
Yapısal Proteinler
Kor 1-191 Nükleokapsid
E1 192-383 Zarf Glikoprotein
E2 384-746 Zarf Glikoprotein
Yapısal olmayan Proteinler
P7 747-809 İyon kanal etkinliği
NS2 810-1026 Proteaz,Transmembran
protein
NS3 1027-1657 Serin proteaz, RNA helikaz
NS4A 1658-1711 NS3’ün serin proteaz aktivitesi
için kofaktör
NS4B 1712-1922 İntraselüler membran
vezikülleri
NS5A 1923-2420 ISDR (İnterferon direnci?)
8 2.1.3.1.1. HCV’nin "Translasyon Olmayan" Bölgeleri
- HCV’nin 5’NCR bölgesi
Hepatit C virüsü genomunun 5’ ucunda bulunan 5’NCR bölgesi 341 nükleotid uzunluğundadır (25). 5’NCR bölgesinde, ORF’nin başlama kodonunun hemen proksimalinde, ribozomlara doğrudan bağlanmaya olanak tanıyan bir “internal ribosomal entry site (IRES)” bölgesi bulunmakadır. HCV 5’NCR’nin yapılan filogenetik ve mutasyon analizleri ile, HCV’nin farklı genotipleri arasında korunan dört büyük yapısal alan (alan I-IV) tespit edilmiştir. 5’NCR’nin ilk 40 nükleotidi, RNA replikasyonunda rol alan, ancak translasyon için gerekli olmayan I alanını oluşturur; dolayısıyla, bu bölgenin işlevi, translasyon için kritik olan 5’NCR’nin geri kalanından farklıdır. Kalan bölümler II-IV, HCV ORF bölgesinin translasyonuna aracılık eden IRES’i oluşturur (36). IRES bölgesi ribozomal 40S alt ünitesine bağlanarak, burada konformasyonel bir değişime neden olmaktadır (35,37).
5’NCR bölgesi, tüm dünyada bulunan HCV suşları arasında en çok benzerlik gösteren korunmuş bölge olması nedeni ile, HCV viremisinin saptanmasında ve kantitasyonunda hedef bölge olarak kullanılmaktadır (38,39).
- 3’NCR bölgesi
3’NCR bölgesi yaklaşık 30 nükleotid içermektedir. Bu bölge, HCV’nin farklı genotiplerinde poly-U ya da poly-A bölümleri ile sonlanmaktadır (35). Poly-U bölgesinden sonra, çok iyi korunmuş olan 98 baz uzunluğunda, 3’-X dizisi adı verilen bir dizi bulunmaktadır. Bu dizinin HCV’nin replikasyonu için gerekli olduğu ve negatif RNA zincirinin sentezinin başlamasında rol oynayabileceği düşünülmektedir (35,37,40).
9 2.1.3.1.2. HCV’nin "Protein Kodlayan" Bölgesi
HCV genomunda, ORF bölgesi büyüklüğü 3010 ile 3033 aminoasit arasında değişen tek bir polipeptid kodlamaktadır. Bu polipeptid sonradan virüs ve konak proteinazları tarafından kesilmekte ve işlevsel olarak 10 farklı protein oluşmaktadır (40). Yapısal proteinler, poliprotein prekürsörlerinden endoplazmik retikulum hücresel sinyal peptidazlarınca, yapısal olmayanlar ise HCV’nin kodladığı iki enzim (viral proteazlar olan NS2-3 ve NS3-4A) aracılığı ile ayrıştırılmaktadır. Poliproteinin N- ucundan itibaren yaklaşık dörtte birlik bölümü virüsün yapısal proteinlerini, kalan kısım ise yapısal olmayan proteinlerini oluşturmaktadır. Virüsün yapısal proteinleri; “Core” (p22), Envelop proteini 1 ve 2 (gp35 ve gp70) ve p7 proteini, yapısal olmayan (nonstructural, NS) proteinleri ise; NS2, NS3, NS4A, NS4B, NS5A, NS5B proteinleridir (35,37,41).
- Yapısal proteinler
HCV’nin yapısal proteinleri olan kor ve zarf glikoproteinleri konağa ait hücresel sinyal peptidazlar kullanılarak ayrılır. Bu olay endoplazmik retikulum membranında gerçekleşir. Kor proteinin 175-191. aminoasitleri arasında kalan karboksi ucu E1 proteini için sinyal peptid görevi görür. Bu kısım sinyal peptid peptidaz tarafından ayrıca kesilir. Olgun kor proteini olarak adlandırılan bu protein endoplazmik retikulum membranından ayrılarak sitozolik lipid damlacıkları ile etileşmek üzere serbest kalır (28).
Kor proteini: Yapısal genlerin kor bölgesi tarafından kodlanan kor proteini, virüsün nükleokapsidini oluşturmaktadır. Birkaç B hücresi epitopu içeren oldukça korunmuş ve çok immünojenik bir proteindir. Kor proteininin HBV transkripsiyonunu ve HIV replikasyonunu baskıladığı, çeşitli hücresel genlerin transkripsiyonunu ve translasyonunu düzenlediği, hücre kültürleri
10 ve hayvan modellerinde onkojenik potansiyele sahip olduğu gösterilmiştir (37,42).
Olgun kor dimerik ve alfa helikal yapıdadır. Yapısal açıdan iki bölümden oluşur. Amino ucunda yer alan ilk bölüm yalaşık 120 aminoasit uzunluğundadır ve RNA’ya bağlanma ile RNA şaperon özellikleri gösterir. Karbosi ucundaki yaklaşık 60 aminoasitten oluşan bölge ise kor proteininin sitozolik lipid damlacıklarına bağlanmasını sağlar. Bu bağlanmanın virüs replikasyonu için gerekli olduğu ve steatozda etkili olduğu düşünülmektedir. Kor proteini mitokondri ve hücre çeirdeğinde de bulunabilmektedir. Mitokondri – kor etkileşimi reaktif oksijen ara ürünlerinin yapımında artışa, Ca+2 bağımlı membran depoarizasyonuna ve apoptoza neden olmaktadır.
Kor proteninin tümör baskılayıcı protein olan PML’yi (promiyelositik lösemi) inaktive etmesiyle ilgili olarak onkojenik potansiyel kazandığı düşünülmektedir (28).
Zarf glikoproteinleri: E1 ve E2 genleri iki adet zarf glikoproteinini kodlamaktadırlar (42). E1 ve E2 proteinlerinin C terminal uçlarında hidrofobik parçalar bulunmaktadır. Bu parçaların membrana tutunmada rolü olduğu düşünülmektedir. E1 ve E2 proteinleri, endoplazmik retikulumda, nonkovalen bağlarla heterodimer kompleks yapılar oluşturur. Dolaşımda LDL ve VLDL’lere bağlanırlar. E1 proteini dört adet korunmuş N-glikolizasyon bölgesi içerir. Glikolizasyon bölgelerinin genotipler arasında korunmuş olması bunların virüs yaşam döngüsünde önemi olduğunu düşündürmektedir (28). HCV sekans karşılaştırmalarında E2’de iki adet çok değişken bölge (Hyper Variable Region, HVR) gösterilmiş, bu bölgeler HVR 1 (aa. 390-410) ve HVR 2 (aa. 474-480) olarak adlandırılmıştır. HCV ile enfekte kişilerde enfeksiyonun seyri sırasında HVR 1’de mutasyonlar oluşmaktadır. Bu bölgedeki yüksek mutasyon oranının konak immün yanıtının seçici baskısı sonucu oluştuğuna inanılmaktadır. E2 bölgesi, virüsü nötralize eden antikorların üretimini sağlamakta, bu da HVR 1 kaçak mutantlı HCV
11 genomlarının immün seçiciliği ile sonuçlanmaktadır. Bu durum, HCV’ye özgü CTL (sitotoksik T lenfosit) ve antikor yanıtının enfeksiyonu iyileştirmede neden etkili olmadığını açıklayabilir. Çünkü varyant genomlar sürekli olarak nötralizasyondan kaçmaktadırlar (37,43,44). E1 ve E2 glikoproteinlerinin ekstrasellüler parçalarının, virüs ile hücre reseptörleri arasındaki etkileşimde önemli bir rol oynadığı düşünülmektedir. E2’ nin lenfosit ve hepatosit gibi çeşitli hücre yüzeylerinde sunulan protein CD 81’e bağlandığı gösterilmiştir (45,46).
- Yapısal olmayan proteinler
Yapısal olmayan proteinler (NS2, NS3, NS4A, NS4B, NS5A ve NS5B) yapısal proteinlerden farklı olarak viral proteazlar olan NS2-3 ve NS3-4A ile kesilirler (47).
p7 proteini: p7 proteini, 63 aminoasitlik bir polipeptid olup, sıklıkla E2 glikoproteini ile birlikte bulunmaktadır. p7 proteininin lipid membranda iyon kanalları oluşturduğu düşünülmektedir (35,48).
NS2 proteini: 212 aminoasitlik hidrofobik bir transmembran proteinidir. NS3 proteinin N-terminal ucu ile NS-3 sistein proteazı meydana getirirler. Hücre kültürlerinde NS2’ nin yokluğunun HCV RNA replikasyonunu engellemediğinin gösterilmesi, viral RNA replikasyonu için gerekli olmadığını düşündürmektedir (49).
NS3 proteini: 631 aminoasitten oluşur ve çok işlevli bir proteindir. N terminal ucunun üçte biri serin proteaz olarak, C-ucunun üçte ikisi ATPaz/RNA helikaz olarak görev yapar (31,50). NS3 serin proteazın proteolitik etkisi NS3/NS4A, NS4A/NS4B, NS4B/NS5A, NS5A/NS5B bileşkelerinde görülmektedir. Bu bazı antiviral ilaçlar için hedeftir (50).
NS4A proteini: 54 aminoasitlik, endoplazmik retikulumda lokalize olan küçük bir proteindir. Viral replikasyonun regülasyonunda rolü olduğu düşünülmektedir. NS4A, NS3 proteinine bağlanarak serin proteaz aktivitesini arttırır ve NS2-NS3 otokatalize proteinazı inhibe eder (51).
12 NS4B proteini: 217 aminoasitlik hidrofobik bir proteindir. Endoplazmik retikulumda lokalize olduğu ve membranla ilişkili olduğu gösterilmiştir. Viral relikasyonun gerçekleştiği “membranöz ağ” olarak adlandırılan bir tür özelleşmiş membran kompartmanı oluşumunu tetikler. NS3, NS4A ve NS4B’nin, NS5A’nın hiperfosforilasyonunu düzenledikleri ve NS5B’nin kodladığı RNA bağımlı RNA Polimeraz (RNA Dependent RNA Polimeraz, RdRp) ile kompleks oluşturdukları gösterilmiştir. Bu etkileşimler NS3, NS4A, NS4B ve NS5B ile birlikte diğer HCV proteinlerinin viral replikasyon için temel oluşturan bir mekanizmanın içinde yer aldıklarını düşündürmektedir (51).
NS5A proteini: 458 aminoasitlik bir fosfoproteindir. Serin aminoasidi üzerinden farklı derecelerde fosforillenmiş p58 ve p56 adı verilen iki formu bulunmaktadır (52). NS5A'nın replikasyon siklusunda rolü olduğu düşünülmektedir. Ayrıca virüsün IFN tedavisine duyarlılığında önemli rolü vardır. NS5A’nın kısa bir bölümündeki dizi polimorfizminin interferona direnci belirlediği saptanmıştır. Bu bölge ISDR (Interferon Sensitivity Determining Region, interferona duyarlılığı belirleme bölgesi) olarak adlandırılmıştır. IFN direncindeki olası mekanizmanın, IFN’nin indüklediği hücresel PKR (protein kinaz) enzimini NS5A’nın inhibe etmesi sonucu olduğu düşünülmektedir (44,53). Ayrıca NS5A’nın çok sayıda hücresel proteinle etkileşime girerek konak hücredeki sinyal ileti yolaklarının kontrolü, apoptozun baskılanması, hücre büyüme ve farklılaşmasının etkilenmesi, lipit metabolizmasının hasara uğratılması ve transkripsiyonun kontrol edilmesi suretiyle birçok konak hücre fonksiyonunu virüsün etkisi altına aldığı bildirilmiştir. NS5A yeni tedavi hedefleri arasında olup günümüzde bazı NS5A inhibitöleri faz II ve faz II deneme aşamasındadır (28).
NS5B proteini: Bu proteinin RNA bağımlı RNA polimeraz aktivitesine sahip olduğu gösterilmiştir. RdRp enzimi negatif ve pozitif iplikli RNA sentezini katalizleyen, viral replikasyonda anahtar rol oynayan bir enzimdir (37). NS5B kodlu RNA bağımlı RNA polimeraz, düzeltme aktivitesinden yoksun, hata eğilimli bir enzimdir. Konak içindeki uygunluğa göre seçilen ve
13 zaman içinde insan popülasyonu içinde farklılaşan mutantlar üretir. Çoğu klinik durumda HCV alt tiplerini karakterize etmek ve enfeksiyonun kökenini belirlemek zordur. NS5B bölgesi, alt tipe özel bir motif içerir ve epidemiyolojik uygulamalar için bir standarttır (54).
2.1.3.2. HCV’nin Yaşam Döngüsü
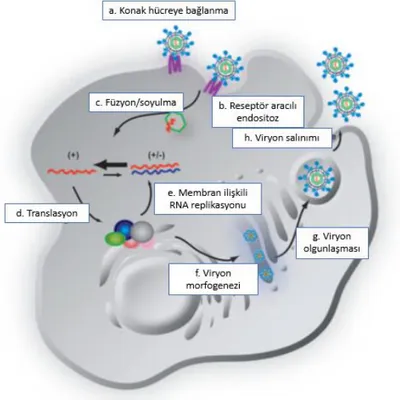
Şekil 3’te HCV’nin yaşam döngüsü şematik olarak görülmektedir.
Şekil 3. HCV yaşam döngüsü şematik diyagramı (55)
a) Ekstraselüler HCV virionları hücre yüzeyinde reseptör molekülleri ile etkileşime girer
b) Düşük pH’lı vesiküle reseptör aracılı endositoz ile geçer
c) HCV glikoprotein aracılı zar füzyonunu takiben, viral RNA sitoplazmada serbest bırakılır
14 d) Genomik RNA, membranöz doku olarak anılan ER benzeri zar yapısı üzerinde 10 olgun HCV proteinine ayrılacak olan tek bir büyük poliproteini üretmek üzere translasyona uğrar
e) Genomik RNA replike olur
f) Yeni sentezlenen RNA’nın bir kısmı nükleokapsidler içine paketlenir ve HCV glikoproteinleri ile ilişkili olarak ER’de tomurcuklanmaya yol açar
g) Viriyonlar hücresel salgı yolağını takip eder ve bu transit sırasında parçacıkların olgunlaşır
h) Olgun viriyonlar hücreden salınır ve yaşam döngüsünü tamamlarlar
HCV RNA’nın virüs ile enfekte olmuş hastaların karaciğerinde bulunması ve deneysel olarak inoküle edilmiş şempanzelerle yapılan çalışmaların sonuçları bu virüsun replikasyonu için temel bölgenin karaciğer olduğunu göstermiştir (56). HCV, ayrıca periferik kan mononükleer hücrelerinde de replike olabilmektedir (57). Ayrıca kemik iliği, lenf nodları, dalak, pankreas, tiroid ve adrenal bezler, primer biliyer epitelyum hücrelerde de HCV replikasyonunun meydana geldiği gösterilmiştir (58).
2.1.3.2.1. HCV Replikasyonu
HCV’nin replikasyonu konusunda bilinenler, virüsün hücre kültüründe üretilmesindeki zorluklar, küçük hayvan modellerinin olmaması, hasta serumunda ve karaciğerinde düşük titrelerde bulunması nedeni ile yetersizdir. HCV replikasyonu hakkında bilgilerin elde edilmesinde flavivirüslerin replikasyon şemasından bazı çıkarımlar yapılmıştır. İlk kez Lohman 1999’da replikon teknolojisine dayalı bir HCV replikasyon modeli tanımlamış ve bu model etkin olarak kullanılmıştır (40).
15 Yıllarca HCV’nin hücre kültüründe üretilmesindeki başarısızlıklar, replikasyonun tam olarak anlaşılabilmesi ile yeni ve ucuz tedavi çalışmaları için engel teşkil etmiştir. 2005’de HCV (JFH1) için in vitro hücre kültür sisteminin geliştirilmesi bu alanda büyük buluş olmuştur (59,60). Fulminan hepatitli Japon hastalardan klonlanan JFH1 viral genom, hücre kültüründe etkili olarak replike olmasının yanı sıra viral partikülleri de üretebilmektedir. HCVcc (hücre kültüründe çoğalan HCV), araştırmacılara HCV’nin replikasyon döngüsünü çalışma olanağı vermiştir.
HCV’nin replikasyon döngüsünde ilk adım, viral partiküllerin hücre reseptörlerine yapışması ve hücre içine virüsün reseptör aracılıklı endositoz ile alınmasıdır. Virüsün hücreye girişinde çeşitli reseptör adayları tanımlanmıştır; CD 81, Glikozaminoglikanlar, SR-BI (Scavenger receptor class B type I), DC-SIGN, L-SIGN, ASGP-R (Asiyaloglikoprotein reseptörü), occludin ve claudin reseptörleri (61,62).
HCV reseptörü olarak en olası aday, tetraspanin CD81’dir (46). CD81 birkaç hücre tipi dışında tüm hücrelerin yüzeyinde bulunur.
Yapılan çalışmalarda, CD81 reseptörüne anti-CD81 antikorları deneysel olarak bağlandığında; HUH-7 hücrelerine ve insan hepatositlerine, HCV’nin girişinin engellendiği rapor edilmiştir (63,64). Ancak yapılan bir başka çalışmada, CD81’in tek başına HCV girişi için yeterli olmadığı Scavenger reseptor B tip 1 (SR-B1) gibi bir kofaktöre ihtiyaç duyduğu gösterilmiştir (65).
HCV partikülü ile etkileştiği gösterilen diğer bir aday, düşük yoğunluklu lipoprotein (LDL) reseptörleridir. Düşük yoğunluklu lipoprotein reseptörü (LDL-R), reseptör aracılıklı endositoz vasıtasıyla hücrelerde lipoprotein giriş çıkışını sağlar. Dolaşımda bulunan HCV’lerin çeşitli oranlarda LDL ile kaplı olmaları nedeniyle HCV’nin LDL-R’yi de hücreye girişte reseptör olarak kullanabileceği düşünülmüştür (28).
16 Virüsün hücreye penetrasyonundan sonra genomik RNA sitoplazmada virüs partikülünden kurtulmakta ve viral genom IRES aracılığıyla ribozomlara yönlendirilmektedir. HCV’nin 3’NCR’de bulunan X bölgesi, IRES’e bağımlı gerçekleşen translasyonun etkinliğini arttırmaktadır. Hücresel ve viral proteazlarla katalizlenerek, kotranslasyonel ve posttranslasyonel olarak viral yapısal ve yapısal olmayan proteinler oluşturulurlar. Viral NS2-5B proteinleri ve konakçı proteinlerin endoplazmik retikulumun sitoplazmik yüzeyinde kalıp viral RNA ile birlikte replikaz kompleksini oluşturdukları düşünülmektedir. NS5B RdRp enzimi (+) iplikli viral RNA genomunu kullanarak, (-) iplikli RNA sentezini katalizlemekte, oluşan (-) iplikli RNA’dan virüsün yeni (+) iplikli RNA sentezi gerçekleşmektedir. Viriyonun birleşmesi endoplazmik retikulumda olgunlaşan Kor, E1, E2 proteinlerinin birbirleriyle olan etkileşimlerine bağlıdır. Kor proteini polimerizasyon ile nükleokapsidi oluşturmakta ve virüs endoplazmik retikulumdan tomurcuklanarak zarf kazanmaktadır. Olgunlaşan virüsün golgi cisimciğinde transportu gerçekleşmekte ve ekzositoz yoluyla hücre dışına salınmaktadır (40,61,62).
2.1.4. HCV Epidemiyolojisi, Bulaş Yolları ve Korunma
2015 yılında, HCV enfeksiyonunun küresel prevelansı %1’dir. Doğu Akdeniz Bölgesi en yüksek prevalansa (%2,3) sahip iken, bunu Avrupa Bölgesi (%1,5) izlemektedir. Dünya çapında HCV’ye yakalanmış 185 milyondan fazla insanın bulunduğu tahmin edilmektedir (3). Yetmiş bir milyon bireyin ise kronik hepatit C hastası olduğu tahmin edilmektedir (4).
Türkiye’de Türk Kızılayı Kan Merkezi’ne 2008-2012 yılları arasında başvuran 4,510,207 donörde anti-HCV pozitiflik oranları incelendiğinde %0,02 - %0,04 arasında olduğu gözlenmiştir (66). Tüm ülkede hemodiyaliz veya periton diyalizi tedavisi gören ve transplantasyon yapılan hastalarda 2010 yılında yapılan bir değerlendirmede anti-HCV pozitiflikleri hemodiyaliz
17 hastalarında %8.5, periton diyalizi hastalarında %4.5, böbrek transplantasyon hastalarında ise %8.4 olarak bildirilmiştir (67).
HCV bulaşı ile ilişkili başlıca risk faktörleri şunlardır: -Kontamine kan ve kan ürünleri transfüzyonu,
-Geçirilen cerrahi operasyonlar,
-Enfekte donörden solid organ transplantasyonu, -İntravenöz uyuşturucu kullanımı,
-Güvenli olmayan tedavi amaçlı yapılan enjeksiyonlar, -İğne batması gibi mesleki temaslar,
-Enfekte anneden bebeğe bulaş, -Hemodiyaliz hastası,
-HIV pozitif hasta,
- Enfekte partnerle ve çoklu partnerle cinsel ilişki (68,69). 2.1.5. HCV Enfeksiyonunda Patogenez ve İmmun Yanıt
HCV enfeksiyonunun patogenezinde hala açıklanamamış birçok nokta bulunmaktadır. Virüsün hücre kültürü modelinin bulunmaması nedeniyle direk sitopatojenitesi güvenilir biçimde test edilememektedir. Virüsün direk olarak sitopatik etki göstermediği, patogenezde başlıca rolü HCV’ye karşı oluşan immün yanıtın oynadığı düşünülmektedir (70).
HCV enfeksiyonunda iyileşmeyi ya da kronik hastalığın oluşmasını belirleyen mekanizmanın ne olduğu tam olarak anlaşılamamıştır. Anti viral nötralizan antikorlar virüsle birlikte bulunmakta, ancak onu nötralize
18 edememektedirler. Ayrıca zamanla türümsülerin gelişimi de HCV’nin immün yanıttan kaçmasını sağlayan en önemli faktördür. Enfeksiyonun kronikleşmesinde; virüse karşı yetersiz immün yanıt oluşması ve virüsün oluşan yanıttan kaçması sorumlu tutulmaktadır (71).
HCV’ye karşı oluşan humoral ve hücresel immün yanıtlar poliklonal ve mültispesifiktir. Sitokin oluşturan CD4+ T Helper ve CD8+ Sitotoksik T (CTL) hücreleri virüs replikasyonunun baskılanmasında etkili olduğu gibi, karaciğer hasarının oluşmasında önemli rol oynamaktadırlar (71,72).
Virüse karşı immün sistemin savunmasında görev alan ilk hücreler, NK (Natural Killer) hücreleridir. Aktive olmuş NK hücreleri büyük miktarlarda sitokin (IFN-γ, IL-4) salınımına neden olur ve virüsle enfekte hücreye karşı spesifik immün yanıt gelişimini tetikler. Ayrıca perforin ve/veya Fas Ligand aracılı sitotoksik etki gösterirler (61).
Akut olgularda, viral yapısal antijenlere karşı belirgin CD4+ T hücre proliferasyonu gelişmektedir. Bu hücreler IL-2 ve IFN-γ salgılarlar. Bununla Th1 yanıtı ortaya çıkmış olur. Th1 yanıtı, sitotoksik T lenfosit (CTL) oluşumunu ve NK hücrelerinin aktivasyonunu tetikler. Virüsün temizlenmesinde Th1 yanıtının başlattığı antiviral mekanizmalar etkilidir. HCV’ye spesifik CTL aktivitesi periferik dolaşımdan çok karaciğerde bulunur. Karaciğerdeki CTL yanıtları erken dönemde multispesifik ve güçlü olarak oluşursa virüs temizlenebilmektedir. Ancak aynı zamanda CTL yanıtı virüsle enfekte hücrelerin apopitozunu tetikleyerek hepatosit hasarına da neden olur. CTL’ler karaciğer hasarına neden olsalar da viral replikasyonu sınırlamada çok önemlidirler (72). Kronik enfeksiyonda ise CD4+ T hücreleri, CD8+ T hücre yanıtlarının sürekliliği için gereklidir. (73).
HCV’nin E2 zarf bölgesindeki epitoplara karşı gelişen antikor yanıtı nötralizan özelliktedir. Bu nötralizan antikorlar serbest virüsü nötralize eder, hücreye girişini engeller ve enfekte hücrelerin antikor aracılıklı sitolizini
19 sağlarlar. HCV, E2 proteini ile CD81’e bağlanarak hücreye girmektedir. Nötralizan antikorlar için hedef bölge Hyper Variable Region-1 (HVR-1)’dir. Bu bölgenin değişkenliği antikorların koruyucu etkisini engeller. İyileşen hastalarda HVR-1’e karşı, kronikleşen olgulara nazaran daha hızlı bir antikor yanıtı oluşmaktadır. Anti HVR-1 immün yanıtı doğal enfeksiyondan sonra, core ve NS proteinlerine karşı oluşmuş antikor titrelerinin düşmesinden daha hızlı bir şekilde düşmektedir. Bu durum, iyileşmiş HCV enfeksiyonunun tam bir bağışıklık bırakmamasının sebebi olarak gösterilebilir. Erken oluşan anti HVR-1 yanıtının HCV enfeksiyonunun iyileşmesi ile anlamlı bir ilişkisinin olduğu gösterilmiştir (72).
2.1.6. Viral Genotipler
Choo ve arkadaşları tarafından 1991’de HCV genomu belirlendikten sonra dünyanın farklı bölgelerinde birçok HCV tipleri elde edilmiş ve sıralanmıştır (27,74). Genom dizileri belirlenmiş HCV suşları incelendiğinde, virüsün genomu boyunca hemen hemen tüm bölgeleri kapsayan, DNA ya da protein dizisinde benzerlikleri fark edilmiş ve bunlar grup ve subgruplar halinde sınıflandırmıştır. Bu sınıflandırma genotiplerin ortaya çıkmasına yol açmıştır. Genotipler Arap rakamları ile (1,2,3,..), alttipleri ise küçük Latin harfleri ile (a,b,c,..) gösterilmektedir (11). Farklı genotiplere ait HCV türleri, nükleotid alanlarının %30-35'inde farklılık gösterir. HCV her bir genotip içinde, ayrıca onaylanmış 67 ve geçici 20 alt tipte sınıflandırılmıştır. Aynı alt türe ait suşlar, nükleotid alanlarının <%15’inde farklılık gösterir (8,9).
HCV genotiplerinden özellikle 1a, 1b, 2a ve 3a alt türlerinin yaygın olarak dünyaya dağılmış olduğu ve yüksek gelirli ülkelerde HCV enfeksiyonlarının büyük bir bölümünü oluşturduğu tespit edilmiştir (10). Ülkemizde de dünya geneliyle benzer şekilde genotip 1b’nin en yaygın genotip olduğu gösterilmiştir (11–20).
20 Genotip 7 ilk kez Kanada’da Afrikalı bir hastadan izole edilmiştir. Şimdilik sadece aynı bölgede birkaç vakada varlığı gösterilmiştir (75).
İlk kez 2002 yılında Saint Petersburg’da doğal bir HCV rekombinant formu tanımlanmıştır. Şimdiye kadar sadece rekombinant HCV genotip 2k/1b virüsleri aktif olarak dolaşımda bulunurken, genotip 2/5, 2b/1b, 2b/1a ve 2i/6p rekombinantları, Avrupa’da, İrlanda, Kıbrıs, Estonya, Almanya ve Fransa’da insanlardan tekli izolatlar halinde gösterilmiştir (1,76).
Viral genotip belirleme enfeksiyonun kaynağını belirlemede ve tedavi modülasyonunda kullanılmaktadır. HCV’ye doğrudan etkili antivirallerin tedavide kullanıma girmesi de genotiplerin ve alt tiplerin doğru belirlenmesinin daha da önemli olmasına sebep olmuştur. Genotip 1b enfeksiyonlarında kronik hepatit ve siroz gelişim oranı daha yüksek, IFN sağaltımına yanıt ise daha az olmaktadır. Genotip 1b ile enfekte karaciğerin nakledildiği olgularda, daha sık ve hızlı olarak akut ve kronik hepatit geliştiği gösterilmiştir. Damar içi uyuşturucu kullananlarda genotip 3 ve 2, kan verilmesi sonrası ve sporadik hepatitlerde ise genotip 1b’nin neden olduğu enfeksiyonlara daha sık rastlanmaktadır (38).
- HCV genotiplerinin coğrafik dağılımı
HCV genotiplerinin coğrafik dağılımının bilinmesi, epidemiyolojik çalışmalar ve risk grupları açısından önem taşımaktadır.
HCV genotip 1 dünya çapında en yaygın olan genotiptir. 83.4 milyon vaka (tüm HCV vakalarının %46.2’si) genotip 1’dir ve bunların yaklaşık üçte biri Doğu Asya’dadır. Genotip 3, bir sonraki en yaygın küresel genotiptir (54.3 milyon, %30.1). Genotip 2, 4 ve 6, tüm vakaların toplam %22.8’inden sorumludur. Genotip 5 geri kalan %1'i içerir. (Şekil 4)
21 Şekil 4. 6 büyük HCV genotipinin coğrafik yaygınlığı (10)
2.1.7. HCV Enfeksiyonunun Kliniği
HCV enfeksiyonu yavaş seyirli bir hastalıktır. HCV ile oluşan akut ve kronik enfeksiyonlarda karaciğer hasarından sorumlu mekanizma halen tam olarak anlaşılamamış ve hastalığın doğal seyri tam olarak çözülememiştir. Doğal bağışıklık sistemi enfeksiyonun başlangıç evresinde kontrol için son derece önemlidir. Viral iyileşme yaygın hücresel immün yanıtla ilişkilidir. HCV enfeksiyonundaki yüksek kronikleşme oranı HCV’nin hızlı mutasyona eğilimi nedeniyle oluşan genetik çeşitlilik sonucu, virüsün immün sistemden kaçmasıyla açıklanmaktadır. Kronik enfeksiyonu olan hastalarda periferik kanda CD4 T lenfositi yanıtının, CD8 lenfositi yanıtına göre daha az olduğu bildirilmiştir. HCV enfeksiyonunun doğal seyrini, konağa ve virüse ait birçok değişken etkilemektedir (77). Şekil 5’te HCV enfeksiyonunun doğal seyri görülmektedir.
22 Şekil 5. Hepatit C virüsü enfeksiyonunun doğal seyri (77)
2.1.7.1. Akut Hepatit C (AHC) Enfeksiyonu
Hepatit C enfeksiyonu, olguların çoğunda anikterik ve subklinik seyreder. Bu nedenle akut dönemde tanımlanması oldukça güçtür. Genellikle ikterik olgular %25’in altındadır. AHC’nin inkübasyon periyodu ortalama 6-8 haftadır. Kan ve kan ürünleri ile bulaşta ise inkübasyon periyodu 2-4 haftaya düşmektedir. AHC’de serum ALT düzeyi genellikle 600 U/l’yi aşmamaktadır. Olguların ortalama %25’inde iyileşme gözlenmekte iken, %25’inde karaciğer hasarı hafif düzeyde kalmakta, yarısında ise ilerleyici bir seyir görülmektedir. İlerleme gözlenen olgularda serum ALT düzeyi sürekli yüksek kalabileceği gibi bazen yükselip bazen de normal sınırlar içerisine inebilmektedir. Bazı olgularda ise serum ALT düzeyi normal sınırlar içerisinde kalsa da histolojik olarak ilerleme görülmektedir. AHC enfeksiyonu sonrası fulminan hepatit gelişimi çok nadirdir (25,62).
23 2.1.7.2. Kronik Hepatit C (KHC) Enfeksiyonu
Akut hepatit C enfeksiyonlarının bir kısmı kendiliğinden iyileşirken önemli bir kısmı ise kronik hepatit C’ye dönüşmektedir. Hepatit C virüsü ile enfekte olan kişilerden %50 ile %80’inde hepatit C enfeksiyonu kronikleşmektedir (78).
KHC tanısı konulan kişilerin yarısından fazlasında tanı ya kan bağışı sırasında ya da kontrol amacıyla yapılan bir tetkikte konulmaktadır. Hastalık dekompanse siroz veya HCC aşamasına gelmemişse, genellikle hastalarda yakınma yoktur. Bazen halsizlik, kuşkulu ağrılar (kas, eklem veya karın sağ üst kadran ağrıları gibi) ve dispepsi gibi yakınmalar olabilmektedir. Fizik muayene veya ultrasonografi bulgusu olarak hepatomegali her zaman saptanamamaktadır. Rastlantısal olarak anti HCV pozitif bulunan olguların bir kısmında aktif HCV enfeksiyonu yoktur. Aktif enfeksiyonu olanların ise hemen tümünde, serum ALT düzeyi kalıcı olarak normal sınırlarda olsa da, karaciğerde histolojik değişiklikler saptanmaktadır. Serum ALT düzeyi geçici de olsa yüksek olan kişilerde, alkole bağlı olduğu düşünülen karaciğer hastalıklarında, Hepatit B ile enfekte kişilerde de mutlaka anti-HCV taraması yapılmalıdır (79).
Sonuç olarak KHC’nin tanısında ve izleminde yakınmaların genellikle olmaması ya da olsa da bu yakınmaların KHC’ye özgü olmaması nedeni ile laboratuvar tetkikleri ve histolojik inceleme önem kazanmaktadır.
2.1.8. HCV Enfeksiyonunda Özgül Mikrobiyolojik Tanı
Hepatit C virüsü enfeksiyonlarında viroloji laboratuvarı tanısal testlerin yapılması ve yorumlanması ile hastaya uygun olan antiviral tedavisine yönlendirilmesi ve izlenmesi konusunda etkin role sahiptir.
Günümüzde, HCV enfeksiyonlarında tanı HCV’ye karşı oluşan antikorları ve HCV antijenlerini saptayan serolojik yöntemler, HCV-RNA’yı saptayan moleküler yöntemler ile konulmaktadır (80).
24 2.1.8.1. Serolojik Yöntemler
-HCV antikor testleri
Hepatit C virüsü antikorlarını tespit etmek için kullanılan HCV EIA (HCV ELISA) ve CIA olmak üzere iki ticari antikor testi vardır.
En yaygın HCV antikor testi HCV EIA veya ELISA'dır. Olumlu bir HCV antikor testi sonucu yalnızca kişinin bir kere hepatit C virüsü ile enfekte olduğunu gösterir; kişinin hepatit C virüsü ile aktif olarak enfekte olup olmadığını öğrenmek için HCV RNA viral yük testi yapılması geremektedir (81).
Kan bankalarında ve klinik mikrobiyoloji laboratuvarlarında tarama amacı ile Anti-HCV antikorlarının saptanması sık kullanılmaktadır. Anti-HCV EIA tarama testleri ile geçirilmiş veya geçirilmekte olan HCV enfeksiyonu saptanabilmektedir (82,83).
HCV ile enfeksiyondan sonra immün sistem tarafından oluşturulan antikorlar, vücut virüsü elimine ettikten sonra kaybolmaz, fakat bu antikorlar tekrar HCV enfeksiyonuna karşı korumada etkisizdir (81).
Günümüze kadar HCV enfeksiyonunda tarama testi olarak kullanılma amacı ile üç kuşak EIA testi geliştirilmiştir. İlk olarak 1989’da geliştirilen 1. kuşak Anti HCV EIA tarama testlerinde rekombinan olarak elde edilmiş HCV genomunun NS4 bölgesinin c100-3 antijeni kullanılmıştır (84). Birinci kuşak Anti-HCV EIA testlerinde duyarlılık %64, pencere dönemi ise en az 3 ay olarak saptanmıştır (85,86).
1991’de 2. kuşak EIA testlerinin geliştirilmesi ile kan transfüzyonu sonrası görülen hepatit olgularının insidansı belirgin olarak azalmıştır (86,87). İkinci kuşak EIA testlerinde NS4 bölgesine ek olarak “core” bölgesinden c22-3 ve NS3 bölgesinden c33c antijenlerinin de test edilmesi ile duyarlılık ve özgüllük arttırılmış, pencere dönemi ise iki aya düşürülmüştür (85,88).
25 1993’de kullanılmaya başlanan 3. kuşak EIA testleri “core”, NS3, NS4 ve NS5 bölgesinden rekombinan veya sentetik antijenler içermektedir. 2. kuşakta kullanılan NS3 ve core bölgesindeki antijenlerin rekonfigürasyonu ile 3. kuşak EIA testlerinde kan donörlerinde duyarlılık ve özgüllük %100’lere ulaşmış, pencere dönemi de 30 güne kadar düşürülmüştür (82,85,89). Birinci, ikinci ve üçüncü kuşak ELISA testlerinde kullanılan antijenler şekil 6’da gösterilmektedir.
Şekil 6. Birinci, ikinci ve üçüncü kuşak ELISA testlerinde kullanılan antijenler
-HCV antijen testleri ve kombine testler
Hepatit C virüsü kor antijeninin saptanmasına yönelik ilk çalışma 1996 yılında Hosseini ve arkadaşları tarafından yapılmıştır. HCV antijen testi, HCV-RNA nın pozitifleşmesinden bir iki gün sonra ve bulaşı izleyen ortalama 27.9 günde pozitifleşir. Bu yüzden kan bankacılığında yararlı olabileceği düşünülmektedir. İlk nesil antijen testleri yeterince duyarlı olmadıkları için bir süre sonra piyasadan kaldırılmıştır. Yeni nesil testlerin ise bazıları sadece kor antijenini saptarken bazıları da (kombine testler) hem antijen hem antikorları saptayabilmektedir. Birçok araştırmada antijen testlerinin HCV-RNA düzeyi ile korele olduğu gösterilmiştir. Karaciğer dokusu ve serumdaki antijen seviyelerinin birbirleriyle ve HCV-RNA düzeyleriyle ilişkili olduğu
26 bildirilmiştir. Yeni kuşak HCV antijen testlerinin viral replikasyonu göstermek, tanı koymak ve antiviral tedavi yanıtını izlemek için rutin kullanıma girmemekle birlikte, kullanılabileceği belirtilmektedir (28,90).
2.1.8.2 Moleküler Yöntemler
Akut ve kronik HCV enfeksiyonlarının tanısında ve kronik HCV hastalarında tedavinin izlenmesinde nükleik asit temelli testler etkin olarak kullanılmaktadır. Aktif HCV enfeksiyonunun gösterilmesinde HCV RNA’nın saptanması altın standart olarak kabul edilmektedir. AHC’de aminotransferazların yükselmesi ve anti-HCV pozitifleşmesinden önce serumda tespit edilebilir. Virüsün vücuda alınmasından 1-3 hafta sonra HCV RNA molekülü saptanabilmektedir (91).
HCV RNA testinin gerekli olduğu durumlar şunlardır (82,91); -Serokonversiyon öncesi akut hepatit enfeksiyonu şüphesi, -İmmün yetmezlikli seronegatif hastalar,
-Belirsiz immunoserolojik test sonuçları, -Tedavi altındaki hastaların monitorizasyonu,
-Yeni doğanların HCV enfeksiyonunun araştırılması.
HCV RNA’yı saptamak için kulanılan iki tip moleküler test vardır. Birincisi, kalitatif yani virüsun var olup olmadığını tespit eden test, ikincisi ise kantitatif yani virüsün miktarını ölçen testtir. Kantitatif test genellikle bir hastanın tedaviye cevap verip vermediğini belirlemek için kullanılır. Yapılan çalışmalarda, kandaki HCV RNA miktarı ile hastalık ilerleme durumu arasında ilişki bulunamamıştır. Geçmişte, Hepatit C virüsü miktarı ölçümü, ‘kopya’ olarak rapor edilirdi. Ancak günümüzde, testlerin farklı dalları arasında ölçümleri standardize etmek için bir girişim ile ‘international unit’ (IU) olarak rapor edilmektedir (81).
27 - Kalitatif NAT:
Bu yöntemler özellikle akut enfeksiyonların tanısı, tedaviye yanıtın incelenmesi ve transfüzyon güvenliğinin sağlanması amacıyla kullanılmaktadır. Sadece viral genomun varlığı veya yokluğunu bildirmekte, sayısal bir değer vermemektedir. Pencere dönemini, üçüncü kuşak EIA taramalarına göre 35 gün kadar kısalttığı bildirilmiştir (28). Kalitatif HCV RNA testleri klasik Polimeraz Zincir Reaksiyonu (PCR), gerçek zamanlı PCR (RT-PCR) ya da Transcription-Mediated Amplification (TMA) tekniğine dayanmaktadır (91).
RT PCR temeline dayanan hedef çoğaltma yönteminde, örnekten izole edilen RNA, reverse transkripsiyon ile cDNA’ya dönüştürülmekte ve PCR ile spesifik hedef bölge çoğaltılmaktadır. Daha sonra çoğaltılmış olan DNA miktarı farklı yöntemlerle saptanmaktadır.
TMA yönteminde reverse transcriptase ve T7 RNA polimeraz enzimleri kullanılmaktadır. Reaksiyon izotermal koşullar altında gerçekleşir ve primerler T7 RNA polimerazın bağlandığı bölgeyi içerirler. Böylece reverse transkripsiyon ile T7 RNA polimerazın kalıbı olan cDNA’lar sentezlenir ve bu da yeni RNA kopyalarının oluşumunu sağlar. Oluşan RNA amplikonları tekrar TMA döngüsüne girer ve sonraki replikasyon siklusu için kalıplar oluşur (92,93).
- Kantitatif NAT:
HCV-RNA düzeylerini belirlemek için kullanılabilecek testler hedef amplifikasyonuna veya sinyal amplifikasyonuna dayalı testlerdir. Dallı zincirli DNA (bDNA) bir sinyal amplifikasyon yöntemidir. Hedefi amplifiye eden testler ise PCR veya TMA tabanlıdır. Bu testler arasında analitik duyarlılık ve dinamik saptama aralığı bakımından farklılıklar bulunmaktadır.
28 Sinyal çoğaltma yöntemi olan bDNA’da nükleik asitler değil onlara bağlanan işaretler çoğaltılmaktadır. Luminesan substratla elde edilen işaretin ölçümü hedef molekül sayısı ile orantılı olmaktadır (94)
Ticari olarak sağlanan tüm HCV RNA testleri, Dünya Sağlık Örgütü’nün genotip 1 HCV içeren 96/790 veya 96/798 standartlarına göre kalibre edilmiştir. Bu testlerde 1 IU/ml karşılığı olan kopya sayısı 0,9 ile 5,2 arasında değişmektedir. Yapılan standardizasyonlara karşın testler arasında farklı sonuçlar alınabilmektedir. Yeni geliştirilen testlerde bile bu sorun olabilmektedir. Örneğin; Qiagen Artus QS-RGQ testi genotip 4 ve 5 virüsleri olduğundan fazla saymaktadır. Genotip 4 virüsleri olduğundan az kantite eden Cobas AmpliPrep/Cobas TaqMan HCV testinin yeni sürümünde bu sorunun giderildiği bildirilmiştir. HCV-RNA testlerinde yaşanabilen bu sorunlar, testlerde kullanılan primer ve/veya probların bağlanma bölgelerindeki baz farklılıklarından kaynaklanmaktadır (28).
2.1.8.3. HCV Genotip Tayininde Kullanılan Testler
HCV genotipleme yöntemlerinde temel amaç HCV’nin genotipini saptayarak, tedavi yöntemini belirleme ve tedavi süresini öngörebilmektir. Yapılmış çalışmalar, tedavi öncesi viral yük ve genotipin saptanması ile kombine tedavinin düzenlenebileceğini göstermektedir. Genotiplemede altın standart, genomun nükleik asit dizi analizinin yapılmasıdır (95). Kullanılmakta olan genotip belirleme yöntemleri temel olarak genotipik ve serolojik yöntemler olarak gruplandırılmaktadır (38,92).
HCV’de yedi filogenetik ana dal (clade) (genotip) mevcuttur. Bu farklı genotipler 70’e yakın alt tipe ayrılmaktadır (9). Farklı HCV alt tipleri farklı risk gruplarını hedef almaktadır ve farklı genetik özelliklere sahip HCV izolatları farklı coğrafik dağılım özellikleri göstermektedir. HCV epidemisinin haritalamasında şu ana kadar yaygın olarak Sanger zincir sonlandırma reaksiyonu ile elde edilen diziler kullanılmıştır. Bu haritalamalar sayesinde