DNA Dizilimlerindeki Protein Kod Bölgelerinin Tanımlanması için Fourier
Tabanlı Filtreleme Yaklaşımı
Fourier-Based Filtering Approach for Identification of Protein- Coding
Regions in DNA Sequences
Bihter DAŞ
Yazılım Mühendisliği Bölümü Fırat Üniversitesi, Elazığ, Türkiye
bihterdas@gmail.com
İbrahim TÜRKOĞLU Bilgisayar Mühendisliği Bölümü Bingöl Üniversitesi, Bingöl, Türkiye
iturkoglu@bingol.edu.tr
Özetçe — Genomik araştırmalarda protein kod bölgelerinin belirlenmesi çok zordur. Üç baz periyotuna sahip bu kod bölgeleri ekson olarak ta adlandırılır. Bu makalenin amacı, bir DNA dizilimindeki eksonların pozisyonlarını Fourier tabanlı filtreleme yaklaşımı kullanarak tahmin etmektir. Bu amaç doğrultusunda insan türünün M8694 genine ait DNA dizilimleri EIIP haritalama tekniği ile sayısal diziye dönüştürülmüştür. Sayısallaştırılmış DNA dizilimlerinin zaman frekans domeni Fourier yöntemi ile elde edilmiş ve antinotch filtresi kullanılarak 3 periyotlu sinyal filtrelenmiştir. Filtrelenmiş sinyalin ekson bölgeleri belirlenen persentil eşik değerine göre başarılı bir şekilde tanımlanmıştır.
Anahtar Kelimeler — DNA dizilimi, Ekson, Antinotch filtresi, Fourier dönüşümü
Abstract — Identification of protein-coding regions is very difficult in genomic research. This coding regions have 3-base periodicity are named as exon. The aim of this paper is to estimate position of exon in the DNA sequence used filter approach based Fourier. For this purpose, DNA sequences of M8694 gene in human species are transformed to numeric sequences by EIIP mapping technique. Time frequency domain of numeric DNA sequences is obtained by Fourier method and 3-period signal is filtered using antinotch filter. Exon regions of filtered signal are defined successfully according to the specified percentile threshold.
Keywords — DNA sequence, Exon, Antinotch filter, Fourier transform
I. GİRİŞ
Genetik özellikler hücre çekirdeğindeki kromozomlarla taşınır. Kromozomlar DNA ve özel proteinlerin birleşmesinden oluşur. Bir DNA’nın yapı birimi nükleotidlerdir. Nükleotidler şeker ve fosfat ve organik bazlardan oluşur. Bu bazlar Adenin (A), Guanin (G), Timin (T), Sitozin (C) dir. Bir nükleotid hangi organik bazı içeriyorsa o bazın ismiyle nitelendirilirler. Protein ve enzimler üretilirken DNA üzerindeki bazların dizilimleri örnek alınarak bu genlere karşılık gelen RNA
kopya dizilimleri çıkarılır. mRNA olarak isimlendirilen bu RNA’lar çıkartılırken bir genin DNA dizilimleri baştan sona tümüyle okunmaz. DNA nın okunmadan atlanan, mRNA ve protein kodlamasına katılmayan bu bölümlerine intron, kodlanan kısımlarına ise ekson adı verilir. Bir gene ait olan DNA diziliminde o dizilimi ekson ve intron olarak sınıflandırmak bir DNA dizi analizinde oldukça önemlidir. [1]
Şekil 1. Bir DNA dizilimi örneği
Bir genin dizi analizinde homoloji (benzerlik) araştırması yani yeni bulunan bir DNA diziliminin diğer tüm dizilimlerle karşılaştırılması ve bunun sonucunda benzerlerdeki veri tabanında ya da literatürde tanımlanmış bazı biyolojik işlevlerin, yeni bulunan dizilime yakıştırılması olarak tanımlanabilir. Bu yöntemle benzerlikler ve protein kodlayan eksonlar araştırılır ve bir genin mutasyona uğrayıp uğramadığı belirlenebilir [2].
II. MATERYALVE YÖNTEM
Sayısal sinyal işleme teknikleri sembolik sinyallere uygulanamaz. Bu yüzden DNA dizilimlerinin sayısal haritalama teknikleri ile sayısal sinyallere dönüştürülmesi gerekir. Bu makalede insan türünün M8694 genine ait baz dizilimleri NCBI veri tabanından alınmış ve daha sonra EIIP sayısal haritalama tekniği ile sayısallaştırılmıştır. Sayısallaştırılmış DNA dizilimlerinin zaman frekans domeni Fourier yöntemi ile elde edilmiş ve antinotch filtresi kullanılarak 3 periyotlu sinyal filtrelenmiştir. Filtrelenen dizilimin enerjisi hesaplanmış ve belirlenen 978-1-4673-7386-9/15/$31.00 ©2015 IEEE
persentil eşik değerine göre enerji diziliminden ekson (protein kod bölgeleri) tespit edilmiştir. Bu eşik değeri bulunurken küçükten büyüğe sıralanmış filtrelenen enerji dizisinin x. elemanı seçilmektedir X değeri ise filtrelenen enerji dizisinin toplam eleman sayısıyla 0.96 değerinin çarpımının yaklaşık değeri alınmasıyla elde edilmektedir. Gerçekleştirilen uygulamanın adımları şekil 2’ de gösterilmektedir.
Şekil 2: Eksonların tespit edilmesi için Fourier dönüşümüne dayalı
filtreleme yaklaşımının akış şeması
A. Verilerin Elde Edilmesi
Bu çalışmada deneysel veriler için National Center for Biotechnology(NCBI) sitesi Gen bankasından insana ait Homo sapiens (Human) M8694 Met geni (Gen Bank No. AH005332.1) verileri alınmıştır. Makaledeki uygulama için giriş verileri olarak 16000 DNA baz dizilimi alınmıştır. Bu baz diziliminde protein kod bölgelerinin (eksonların) ilgili konumları 8604-8719, 10298-10399, 12388-12433, 14134-14199, 15672-15740 değer aralıklarıdır. Bu aralıkların dışındakiler protein kodlanmayan bölgelerdir.
B. DNA Diziliminin Sayısal Haritalama Tekniği ile Sayısallaştırılması
Sinyal işleme uygulamalarında DNA dizilimleri üzerinde çalışabilmek için DNA dizilimlerinin sayısal sinyallere dönüştürülmesi gerekir. Bu dönüştürme işleminde tek boyutlu ve çok boyutlu sayısal haritalama teknikleri kullanılmaktadır. Bu makale çalışmasında tek boyutlu sayısal haritalama tekniklerinden biri olan EIIP tekniği kullanılmaktadır [3,4]. Bu teknikte DNA dizilimindeki her bir nükleotid EIIP temsilindeki yarı değerlik sayısı ile eşleştirilir. A= 0.1260, G=0.0806, C=0.1340, T=0.1335 değerleri verilir. Eğer X(n) dizisinde A, G, C, T için EIIP değerleri yerine koyulursa, oluşan yeni sayısal dizilim Xe(n) bir DNA dizilimi boyunca
serbest elektron enerji dağılımlarını temsil eder.
X(n)=[ A T T G C A T G C] iken Xe(n)=[0.1260 0.1335
0.1335 0.0806 0.1340 0.1260 0.1335 0.0806 0.1340]
C. Kullanılan Filtreleme Metodu
Bu makale çalışmasında protein kod bölgelerini(ekson) belirlemede Fourier tabanlı spektral tahmin yöntemi kullanılmıştır. Eksonların 3-periyotlu davranışı DNA dizilimlerini filtreleme yoluyla çıkarılmıştır. Filtreleme yaklaşımı olarak Antinotch filtresi kullanılmıştır. Antinotch filtre büyüklük cevabı 2π/3 olan keskin tepeden oluşan bir filtredir. Eğer antinotch filtreye giriş olarak ikili işaret dizilimleri olan UA(n), UT(n), UC(n),
UG(n) verilirse protein kod bölgelerinde onlara karşılık
gelen çıkışlar yA(n), yT(n), yC(n), yG(n) dir. [5] Bu
filtrenin passband(bant geçiren) değeri yaklaşık 2π/3’dir. Sayısal filtre kullanılarak özellik hesaplanması
Y(n)=
∑
i∈F
|
y
i(
n)
|
2 F={
A , T , C , G
}
(1)
Çizilmiş olan Y(n) grafiğinde tepeler kod bölgelerini yani eksonları gösterirken, zayıf çıkışlar ise kodlanmayan bölgeleri yani intronları göstermektedir. Bu amaçla DNA segmentlerinde protein kod bölgelerini (eksonları) belirlemede bu özellikten yararlanılmıştır.
Vaidyanathan ve Yoon tarafından önerilen ve eksonları belirlemede kullanılan IRR antinotch filtresi, dar bant geçiren filtredir ve bu bölgede yüksek bir kazanç sağlar. Antinotch filtresinin transfer fonksiyonu [6,7]
H(z)=
1
2
(1−R
2)(1−z
−2)
(1−2 RCosθ z
−1+
R
2Z
−2)
(2) D. Uygulama SonuçlarıEksonlar, bir DNA dizilimde 3 periyotlu baz (kodon) halinde bulunduklarından genomik uygulamalarında eksonların belirlenmesi zordur. Gerçekleştirilen ekson EIIP haritalama tekniği ile dizilimin
sayısallaştırılması
DNA diziliminin Fourier dönüşümü ile zaman-frekans temsili
Antinotch Filtresi tasarlanması
Enerji diziliminde eşik değere göre exonların tahmini Filtrelenmiş dizilimin enerjisinin
hesaplanması
3 periyotlu sinyalin filtrelenmesi DNA dizilimlerinin NCBI veri
tabanından alınması
tespiti uygulamasında, Fourier dönüşümüne dayalı filtreleme yaklaşımı kullanılarak 3 periyotlu DNA diziliminde eksonların hangi baz pozisyonlarda bulunduğu tahmin edilmiştir [8,9]. Daha sonra da tahmin edilen bu pozisyonlar gen bankasından alınan DNA dizilimindeki gerçek ekson pozisyonlarıyla karşılaştırılmış ve doğruluk
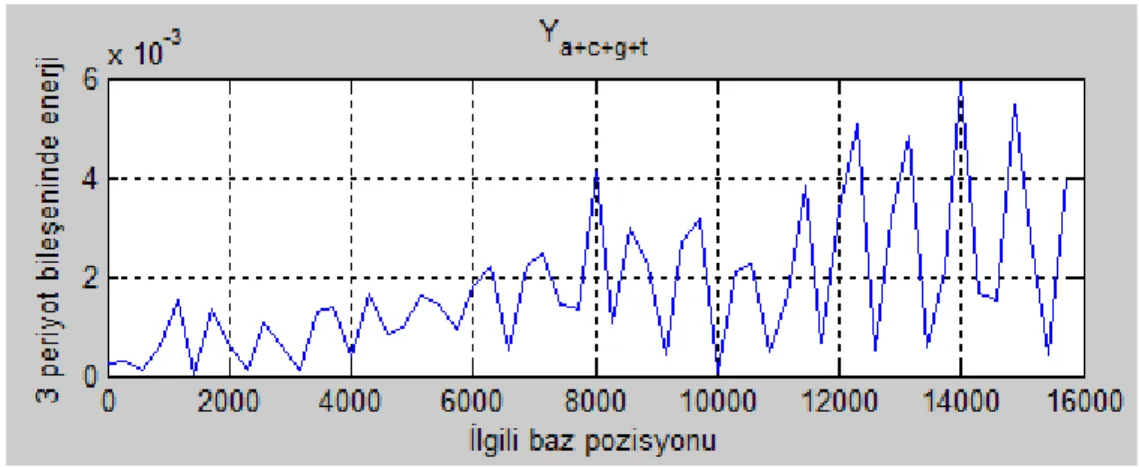
oranları tespit edilmiştir. Şekil 3’de uygulama sonucunda eksonların pozisyonlarını ifade eden güç spektrumları gösterilmiştir. Şekil 3’e göre 870, 1000-1200, 1200-1400, 1500 aralığındaki baz pozisyonları 0.0030 enerji eşik değerini aşmış ve ekson(protein kod bölgesi) olarak dikkate alınmıştır.
Şekil 3: Baz konumlarına göre eksonları ifade eden güç spektrumları Tablo 1’de başlangıçta gen bankasından alınan DNA
dizilimlerindeki ekson bölgelerinin pozisyonları ile yöntemin bulduğu ekson pozisyonları karşılaştırılmış ve yöntemin her ekson pozisyonu için doğrulukları çıkarılmıştır. Yöntemin her pozisyondaki başarı ortalaması yaklaşık %95,30’dur.
Tablo 1: Gen Bankasından Alınan Ekson Pozisyonları İle Filtreleme
Yaklaşımı Sonucu Bulunan Ekson Pozisyonlarının Karşılaştırılması
Gen Bankasında Baz
Pozisyonu(NCBI) Fourier Tabanlı AntinotchFiltre Yaklaşımı Doğruluk Oranı (%)
8604-8719 (115) 8600-8700 (100) 83,47 10298-10399 (101) 10300-10400 (100) 97,029 12388-12433 (45) 12400-12450 (50) 95,55 14134-14199 (65) 14100-14200 (100) 100 15672-15740 (68) 15650-15750 (150) 100 III. SONUÇ
Genomik araştırmaların temel amacı genler tarafından kodlanan özel fonksiyonları belirlemek ve bu genlerin taşıdığı bilgiyi anlamaktır. Genler bir DNA diziliminde ekson ve intron olarak bulunurlar. DNA dizilimlerinden protein kod bölgelerini(ekson) tahmin etmede Fourier tabanlı filtreleme yaklaşımı kullanılmıştır. Bu yaklaşıma göre 16000 baz uzunluğundaki DNA diziliminde ekson olan bölgelerin pozisyonları tahmin edilmiş ve gerçek ekson pozisyonlarıyla karşılaştırılmıştır. Ekson
pozisyonlarını tahmin etmede kullanılan bu yöntemin etkili ve başarılı olduğu görülmüştür.
KAYNAKÇA
[1] Sahu, S. ve Panda, G. “Identification of Protein-Coding Regions in DNA Sequences Using A Time-Frequency Filtering Approach”, Genomic Proteomics&Bioinformatics, October 2010.
[2] Internet:Wikipedia,http://tr.wikipedia.org/wiki/%C4%B0ntron, Erişim Tarihi: Kasım 2014.
[3] Nair, A.S., Pillai, S.S., “A coding measure scheme employing electron-ion interaction pseudo potential (EIIP)”, Journal of Bio-information, Cilt: 1, 197 – 202, October, 2006.
[4] I. Cosic, “Macromolecular Bioactivity: Is it resonant interaction between macromolecules? Theory and Applications”, IEEE Transactions on Biomedical Eng., Cilt: 41, 1101-1114, December 1994.
[5] Hota, M. ve Srivastava, V. “Identification of protein coding regions using antinotch filter”, Digital Signal Processing (22), 869-877, June, 2012.
[6] Vaidyanathan, P. P. ve Yoon, B.-J. The role of signal-processing concepts in genomics and proteomics, J. Franklin Inst. 341 (2004) 111– 135 (Special Issue on Genomics).
[7] Vaidyanathan, P.P. ve Yoon, B.-J. Gene and exon prediction using allpass-based filters, in: Workshop on Genomic Signal Process. Stat., Raleigh, NC, 2002.
[8] Mena-Chalco, J. ,Carrer, H., Zana, Y., Cesar, R. M. Identification of protein coding regions using the modified Gabor-wavelet transform, IEEE/ACM Trans. Comput.Biol. Bioinform. 5 (2008) 198–207
[9] Kotlar, D., Levner, Y. Gene prediction by spectral rotation measure: A new method for identifying protein-coding regions, Genome Res. 13 (2003) 1930–1937.
[10] Daş, B., Türkoğlu, İ., "DNA Dizilimlerindeki Nükleotit Çiftlerinin Frekans Değerlerine Göre Farklı Sınıflandırma Yöntemleri ile Karşılaştırılması", Tıp Teknolojileri Ulusal Kongresi (TIPTEKNO'2014), s.191-194, 25-27 Eylül 2014, Kapadokya.
[11] Daş, B., Türkoğlu, İ., "DNA Dizilimlerinin Sınıflandırılmasında Karar Ağacı Algoritmalarının Karşılaştırılması", Elektrik – Elektronik – Bilgisayar ve Biyomedikal Mühendisliği Sempozyumu (ELECO 2014), s.381-383, 27-29 Kasım 2014, Bursa.
[12] Ramachandran, P., Lu, W.-S., Antoniou, A. Location of exons in DNA sequences using digital filters, in: Proceedings of IEEE ISCAS, pp. 2337–2340, 2009.
[13] Akhtar, M., Epps, J., Ambikairajah, E., Time and frequency domain methods for gene and exon prediction in eukaryotes, in: Proc. IEEE ICASSP, pp. 573–576, 2007.
[14] Abo-Zahhad, M., Ahmed, S. M., Abd-Elrahman, A.S., “Genomic Analysis and Classification of Exon and Intron Sequences Using DNA Numerical Mapping Techniques”, International Journal Information Technology and Computer Science, Cilt: 8, 22-36, 2012.
[15] Hota, MK., Srivastava, VK., “DSP technique for gene and exon prediction taking complex indicator sequence”, Proc. IEEE TENCON, 1-6, 2008.