Meme Kanserinde CerbB2 Tümör Hücrelerinin
SÕnÕflandÕrÕlmasÕ için Derin Ö÷renme TabanlÕ Bir
YaklaúÕm
A Deep Learning Based Approach for Classification
of CerbB2 Tumor Cells in Breast Cancer
Gözde A. Tataro÷lu
1, AnÕl Genç
2, Kaan A. KabakçÕ
1, Abdulkerim Çapar
1, B. U÷ur Töreyin
1, HazÕm K. Ekenel
2ølknur Türkmen
3, AslÕ ÇakÕr
31
Biliúim Enstitüsü, østanbul Teknik Üniversitesi {tataroglu15, kabakci, capar, toreyin}@itu.edu.tr
2
Bilgisayar Mühendisli÷i Bölümü, østanbul Teknik Üniversitesi {genca16, ekenel}@itu.edu.tr
3Patoloji Anabilim DalÕ, østanbul Medipol Üniversitesi {iturkmen, acakir}@medipol.edu.tr
østanbul, TÜRKøYE
Özetçe— Bu çalÕúmada, meme kanserinde CerbB2 tümör
hücre skorlarÕnÕn derin ö÷renme modellerine dayalÕ olarak sÕnÕflandÕrÕlmasÕna yönelik, özgün bir yaklaúÕm önerilmektedir. Bildirinin bir di÷er katkÕsÕ da, özgün meme kanseri dokusundan veri kümesinin oluúturulmasÕdÕr. Derin ö÷renme modellerinin e÷itilmesi, do÷rulanmasÕ ve sÕnanmasÕ amacÕyla, doku örnek görüntüleri üzerinden hücre parçalarÕ oluúturulmuútur. Oluúturulan hücre parçalarÕna iliúkin CerbB2 tümör skorlarÕ, evriúimsel sinir a÷larÕ (ESA) yardÕmÕyla yüksek baúarÕmda sÕnÕflandÕrÕlmÕútÕr.
Anahtar Kelimeler — CerbB2 tümör iúaretleyici; tümör; Evriúimsel Sinir A÷larÕ (ESA); skor; sÕnÕflandÕrma.
Abstract— This study proposes a unique approach to classify
CerbB2 tumor cell scores in breast cancer based on deep learning models. Another contribution of the study is the creation of a dataset from original breast cancer tissues. On the purpose of training, validating and testing with deep learning models cell fragments were generated from sample tissue images. CerbB2 tumor scores were generated for the cell fragments were classified with high performance by the aid of convolutional neural networks (CNN).
Keywords — CerbB2 marker; tumor; Convolutional Neural Networks (CNN); score; classification.
I. GøRøù
Günümüzde, meme kanseri, yaygÕnlaúan bir hastalÕk olarak incelenip, bilimsel çalÕúmalara sÕklÕkla konu olmaktadÕr. YaygÕnlaúan bu kanser türünde, özellikle tedavi sürecinde baúarÕyÕ arttÕrmaya yönelik olarak disiplinler arasÕ birçok çalÕúma ortaya çÕkmaktadÕr. Bu amaç do÷rultusunda da, hedefe yönelik tedavi (targeted therapy/treatment) yaklaúÕmlarÕ son yÕllarda yaygÕnlaúmaktadÕr. Hedefe yönelik tedavi amacÕyla epidermal büyüme reseptörü (EGFR) ailesine ait bir protein olan
CerbB2 tümör iúaretleyicisi kullanÕlmaktadÕr [1, 2]. Meme dokularÕnda, mutasyona u÷ramÕú CerbB2 tümörlü hücreler, hücre çeperinde bulunan CerbB2 proteininin artÕúÕyla tespit edilmektedir [3]. Protein artÕúÕnÕn görünür kÕlÕnÕp patologlar tarafÕndan de÷erlendirilebilmesi için, FISH (Fluorescent in Situ Hybridization) ve ømmuno Histo Kimya (øHK) tabanlÕ olmak üzere iki farklÕ boyama tekni÷i kullanÕlmaktadÕr [4, 5]. Bu tekniklerden, zaman ve maliyet açÕsÕndan daha uygun olan ve ASCO / CAP 2013 önerilerine göre dört dereceli bir skor ölçe÷i içeren øHK yaklaúÕmÕ, meme kanseri tedavi sürecinin belirlenmesinde sÕklÕkla kullanÕlmaktadÕr [6] (bkz. ùekil 1).
Doku örneklerine skor de÷erlerinin atanmasÕ sürecinde, patologlara yardÕmcÕ olmak amacÕyla görüntü iúleme yöntemleri kullanÕlmaktadÕr [7, 8]. Bu yöntemlerden birçok kanser türünde oldu÷u gibi, meme kanseri teúhisinde de yararlanÕlmaktadÕr [9, 10]. Bu ba÷lamda, son yÕllarda geliútirilen yaklaúÕmlardan biri olan derin ö÷renmeye dayalÕ tekniklerle, kanserli doku çözümlemesine yönelik çalÕúmalarÕn arttÕ÷Õ gözlenmektedir [11, 12].
Patologlardan ve makine ö÷renmesi araútÕrmacÕlarÕndan oluúan bir ekip tarafÕndan yürütülen bu çalÕúmada, literatürdeki di÷er yöntemlerden farklÕ olarak, meme kanserinin tedavi sürecinin belirlenmesinde ihtiyaç duyulan CerbB2 tümörlü hücre skorlarÕ, derin ö÷renme modellerine dayalÕ bir yaklaúÕmla sÕnÕflandÕrÕlmÕútÕr. Bu amaçla, öncelikle, patolojik doku örnekleri üzerinden kesitler alÕnarak özgün bir veri kümesi elde edilmiútir. Bu veri kümesinden de hücre kesitleri oluúturulmuú, derin ö÷renme modelleriyle e÷itilmiú ve CerbB2 tümörleri sÕnÕflandÕrÕlmÕútÕr.
Bu çalÕúmanÕn ikinci bölümünde, oluúturulan veri kümesi hakkÕnda ayrÕntÕlÕ bilgi verilmiú olup, üçüncü bölümünde ise Evriúimsel Sinir A÷larÕ (ESA) ile skorlama anlatÕlmÕú ve bildiri, deneysel sonuçlarÕn tartÕúÕlmasÕyla sonlandÕrÕlmÕútÕr.
ùekil 1. Kanserli meme doku örnekleri (a) Skor 0, (b) Skor 1, (c) Skor
2 ve (d) Skor 3. Skorlar arttÕkça, boya yo÷unlu÷u artmaktadÕr. Yo÷un ve düzgün çevresel boyaya sahip Skor 3 tümörleri, patologlar tarafÕndan, cerbB2
proteinine yönelik tedavi (targeted treatment) için uygun kabul edilir.
II. HÜCREPARÇALARININOLUùTURULMASI Öncelikle, bilgisayar ortamÕnda incelenecek olan øHK doku örnekleri, dijital mikroskop sistemi ile (1,4 megapiksel sensör çözünürlü÷ü ile) sayÕsallaútÕrÕlmÕútÕr. Mikroskobun, mozaik görüntü alma aracÕ da, görüntü alanÕndan daha büyük doku bölgelerinin panoramik görüntülerini yakalamak için kullanÕlmÕú ve veri kümesi elde edilmiútir
Elde edilen doku görüntüleri, hücre bölgelerinin tespiti için, renk ayrÕútÕrma yöntemiyle, hücre çekirdeklerini boyayan hematoksilen (H-Mavi) ve hücre çeperlerini boyayan diaminobenzidin (DAB-Kahverengi) olarak renk kanallarÕna ayrÕútÕrÕlmÕútÕr [13]. H-mavi kanal görüntüsüne çok seviyeli eúikleme uygulanarak hücre merkez koordinatlarÕ tespit edilmiútir [14]. AyrÕca ilgili çalÕúmalarda FISH görüntülerine uygulanan çok seviyeli eúikleme yöntemi, mevcut çalÕúmada da øHK görüntülerine uygulanmÕútÕr.
Tespit edilen koordinatlarÕ merkez kabul eden, 84x84 piksel çözünürlü÷ündeki hücre parçalarÕ, skoru önceden patologlar tarafÕndan belirlenmiú 208 adet mikroskobik doku görüntüsünden kesilerek oluúturulmuútur. Böylece, derin ö÷renme modellerinin e÷itilmesi, do÷rulanmasÕ ve sÕnanmasÕ amacÕyla toplam 50990 hücre parçasÕ elde edilmiútir. Bu parçalardan, 12700’ü Skor 0 tipi, 14790’Õ Skor 1 tipi, 12600’ü Skor 2 tipi ve 10900’ü ise Skor 3 tipi hücre parçasÕndan meydana gelmiútir. Oluúturulan hücre parçalarÕna ait bazÕ örnekler Tablo 1’de verilmiútir.
III. ESAøLESKORLAMA
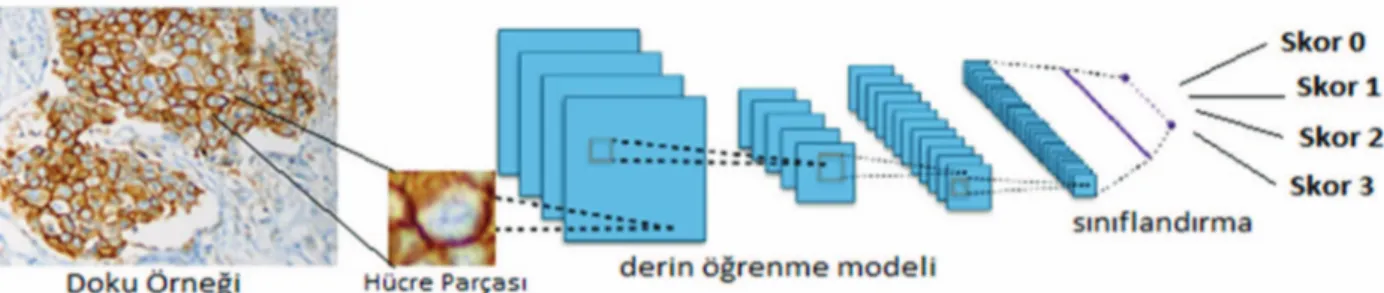
Hücreler kesilip veri kümesi hazÕrlandÕktan sonra, veriler, evriúimsel sinir a÷larÕ (ESA) kullanÕlarak, ùekil 2’deki gibi dört farklÕ skor tipine ait sÕnÕfta yüksek baúarÕm oranlarÕnda e÷itilmiú ve CerbB2 tümör hücre parçalarÕnÕn skor sÕnÕflandÕrÕlmasÕ yapÕlmÕútÕr [15]. Derin ö÷renme modellerinin e÷itilmesi, do÷rulanmasÕ ve sÕnanmasÕ, derin ö÷renme modelleri için bir geliútirme ortamÕ olan Caffe platformunda, AlexNet ve VGG-16 mimarileri kullanÕlarak yapÕlmÕútÕr [16-18].
Tablo 1. Veri kümesindeki her bir skor türü için kesilen bazÕ hücre parçalarÕ [6].
SÕnÕflandÕrma aúamasÕnda kullanÕlan evriúimsel sinir a÷larÕyla kayÕp (loss) fonksiyon katmanÕ kullanÕlmÕútÕr. Çünkü kayÕp fonksiyonlarÕ, etiketlenmek istenen veri üzerindeki yanlÕú tahminler kullanarak, sinir a÷ÕnÕ, do÷ru bir úekilde yönlendirmeye çalÕúmaktadÕr [19]. Bu sebeple, kayÕp katmanÕnÕn kararlarÕnÕ oluúturmak için kullanÕlan denklemin tüm sonuç üzerinde etkisi olmaktadÕr. ÇalÕúmadaki sÕnÕf sayÕsÕnÕn da az olmasÕ sebebiyle, kullanÕlan mimarilerin baúarÕsÕ arttÕrÕlmaya çalÕúÕlmÕú ve model oluúturulurken, evriúimsel sinir a÷larÕnÕn son katmanÕnda kullanÕlan “Softmax” kayÕp fonksiyonunun yanÕ sÕra “Hinge” kayÕp fonksiyonundan da yararlanÕlmÕútÕr [20,21].
IV. DENEYSELSONUÇLAR
Bu bölümde, derin ö÷renme mimarileri kullanÕlarak yapÕlmÕú çalÕúmalarÕn e÷itim, do÷rulama, sÕnama sonuçlarÕnÕn baúarÕm tablosu ve her mimari türüne ait karÕúÕklÕk matrisleri, skor tipleri üzerinden paylaúÕlmaktadÕr.
Tablo 2, AlexNet, VGG-16 mimarilerinde Softmax, Hinge kayÕp fonksiyonlarÕ ve Destek Vektör Makineleri (DVM) [22] kullanÕlarak yapÕlan deneylerin sonuçlarÕnÕ göstermektedir. Bu sonuçlara göre, e÷itim verisinin ortalama %99 oranÕnda ö÷renilmiú oldu÷u gözlemlenirken, sÕnama sonuçlarÕnÕn %90’Õn üzerinde kaldÕ÷Õ görülmektedir. SÕnama baúarÕmÕnÕn en yüksek oldu÷u mimari, Hinge loss kullanÕmÕyla VGG-16 olup yaklaúÕk %96 sÕnÕflandÕrma baúarÕsÕ göstermiútir. AlexNet’te Hinge loss kullanÕmÕnÕn ise test sonuçlarÕnÕ %2 oranÕnda düúürdü÷ü görülmüútür. AyrÕca e÷itim ve do÷rulama baúarÕsÕ arasÕndaki farktan yola çÕkarak klasik AlexNet mimarisinin e÷itim verisi üzerinde aúÕrÕ uyum sa÷larken, klasik VGG-16 mimarisinde böyle bir durum yaúanmadÕ÷Õ görülmektedir. Bu durum, kayÕp fonksiyonlarÕnÕn (Softmax, Hinge) derin ö÷renme modelleri üzerinde etkisi oldu÷unu göstererek, VGG-16 mimarisinin baúarÕmÕnÕ Softmax kullanÕmÕna göre yaklaúÕk %1 oranÕnda arttÕrmÕútÕr. Derin ö÷renme mimarileri dÕúÕnda, DVM e÷itimlerinin sonuçlarÕ da Tablo 2’de gözlemlenmiútir. Testler, kayÕp fonksiyonlarÕnÕn ö÷renme modelleri üzerindeki etkisini gözlemlemek adÕna
ùekil 2. Doku örne÷inden hücresel parçanÕn alÕnÕp derin ö÷renme modelleriyle e÷itilmesi sonucu yapÕlan sÕnÕflandÕrmanÕn genel gösterimi.
Tablo 2. KullanÕlan derin ö÷renme mimarilerine ait baúarÕm sonuçlarÕ.
VGG-16 mimarisinden iki farklÕ öznitelik çÕkarÕmÕyla yapÕlmÕútÕr. Burada dikkat edilmesi gereken bir yaklaúÕm da úudur: DVM’ de kullanÕlan öznitelikler, en yüksek sÕnÕflandÕrma baúarÕsÕna sahip olmasÕ nedeniyle sadece VGG-16 mimarisinden elde edilmiútir. Bu öznitelikler yüksek boyutlu oldu÷undan, DVM modelini e÷itirken bu boyut sorununu önlemek amacÕyla basitçe lineer çekirdek düzenlemesi kullanÕlmÕútÕr. Tablo 2’de verilen sÕnama baúarÕmlarÕnÕ her sÕnÕf için daha ayrÕntÕlÕ bir úekilde göstermek amacÕyla, 4 ayrÕ mimari üzerinden elde edilen karÕúÕklÕk matrisleri ùekil 3’te sunulmaktadÕr.
KarÕúÕklÕk matrislerinde, sÕnÕflarÕn etiketleri, daha önceden anlatÕlan hücre skorlarÕ ile aynÕdÕr. Matrisler üzerinde koyu renkle gösterilen alanlar, ilgili satÕr ve sütundaki skor tipine ait do÷ru tahmin oranlarÕnÕ gösterirken, açÕk koyuluktaki alanlar di÷er skor tipleriyle yapÕlan düúük oranlardaki karÕúÕklÕk oranlarÕnÕ göstermektedir. KarÕúÕklÕk matrislerine bakÕldÕ÷Õnda dikkat çeken di÷er bir nokta da, daha çok komúu hücre skorlarÕnÕn birbiri ile karÕútÕrÕldÕ÷ÕdÕr. Bu da, hücre skorlarÕ arasÕndaki fark arttÕkça, ilgili iki sÕnÕfÕn birbiriyle karÕútÕrÕlma olasÕlÕ÷ÕnÕn tümör bölgelerinin da÷ÕlÕm de÷iúimiyle düútü÷ünü göstermektedir.
V. SONUÇ VE TARTIùMA
Bu çalÕúmada, meme kanserinde CerbB2 tümörlerinin skor sÕnÕflandÕrÕlmasÕ, derin ö÷renme modelleri ile uygulanmÕú ve özellikle VGG-16 mimarisiyle iyi derecede test ve e÷itim baúarÕsÕ gözlemlenmiútir. Hinge kayÕp fonksiyonunun kullanÕmÕ da çalÕúmayÕ çeúitlendirmiú ve bu baúarÕyÕ arttÕrmÕútÕr. Böylelikle, kayÕp fonksiyonlarÕnÕn etkisinin, sadece veri kümesine ba÷lÕ de÷il, ayrÕca kullanÕlan derin ö÷renme mimarisine de büyük ölçüde ba÷lÕ oldu÷u gözlenmiútir. Elde edilen karÕúÕklÕk matrisleri incelendi÷inde, Skor 0 ile Skor 2’nin di÷erlerine göre daha yüksek oranda karÕútÕrÕldÕ÷Õ gözlenmiútir. Oluúturulan veri kümesi incelendi÷inde, bu karÕútÕrmanÕn nedeninin Skor 0 bölgelerinde Skor 2 tipine benzeyen tümör kÕsÕmlarÕnÕn da bulundu÷unun gözlemlenmesidir. Fakat bu
karÕúÕklÕk, hücre kesitinin nihai skor türünü de÷iútirmeyecek oranda düúük bir yüzdeli÷e sahiptir.
ùekil 3. KarÕúÕklÕk matrisleri sÕrasÕyla: (a) Softmax kayÕp fonksiyonu ile
AlexNet, (b) Hinge kayÕp fonksiyonu ile AlexNet (c) Softmax kayÕp fonksiyonu ile VGG-16 (d) Hinge kayÕp fonksiyonu ile VGG-16 mimarilerinin
her skor tipi için test sonuçlarÕnÕ göstermektedir.
Mevcut derin ö÷renme modelleriyle elde edilen sÕnÕflandÕrma çalÕúmasÕndaki baúarÕnÕn, tüm bir patolojik slayt görüntüsü üzerinden de elde edilmesi amaçlanmaktadÕr. Bu amaç do÷rultusunda, çalÕúmanÕn devamÕ niteli÷inde, slayt görüntüleri üzerinden ilgili tümör bölgelerinin, evriúimsel sinir a÷larÕ yardÕmÕyla otomatik olarak saptanmasÕna yönelik hücresel ve bölgesel çalÕúmalarÕn yürütülmesi öngörülmektedir.
TEùEKKÜR
Bu çalÕúma, 113E067 numaralÕ TÜBøTAK projesi ve AB 7. Çerçeve ProgramÕ Marie Curie FP7 entegrasyon projesi tarafÕndan desteklenmiútir. AyrÕca çalÕúmada kullanÕlan doku örneklerini sa÷layan Medipol Hastanesi’ne ve hastane bünyesinde görev alan patologlarÕmÕza teúekkürlerimizi sunarÕz. Son olarak, patolojik verilerin görüntülenmesi konusunda geliútirdikleri teknolojikleri kullanÕmÕmÕza sunan Argenit FirmasÕna da teúekkür ederiz.
KAYNAKLAR
[1] L. Garrison and D. Veenstra, "The Economic Value of Innovative Treatments over the Product Life Cycle: The Case of Targeted
Trastuzumab Therapy for Breast Cancer", Value in Health, cilt 12, sayÕ. 8, pp. 1118-1123, 2009.
[2] R. Bhargava, W. Gerald, A. Li, Q. Pan, P. Lal, M. Ladanyi and B. Chen, "EGFR gene amplification in breast cancer: correlation with epidermal growth factor receptor mRNA and protein expression and HER-2 status and absence of EGFR-activating mutations", Modern Pathology, cilt. 18, sayÕ. 8, pp. 1027-1033, 2005.
[3] Cobleigh, Melody A., et al. "Multinational study of the efficacy and safety of humanized anti-CERBB2 monoclonal antibody in women who have CERBB2-overexpressing metastatic breast cancer that has progressed after chemotherapy for metastatic disease." Journal of Clinical Oncology 17.9, 2639-2639, 1999.
[4] Ramos-Vara, J. A., and M. A. Miller. "When Tissue Antigens and Antibodies Get Along Revisiting the Technical Aspects of Immunohistochemistry—The Red, Brown, and Blue Technique." Veterinary Pathology Online 51.1, 42-87, 2014.
[5] Johnson, Kirby L., Dong-Kai Zhen, and Diana W. Bianchi. "The use of fluorescence in situ hybridization (FISH) on paraffin-embedded tissue sections for the study of microchimerism." Biotechniques 29.6, 1220-1224, 2000.
[6] Wolff, Antonio C., et al. "Recommendations for human epidermal growth factor receptor 2 testing in breast cancer: American Society of Clinical Oncology/College of American Pathologists clinical practice guideline update." Journal of clinical oncology 31.31, 3997-4013, 2013.
[7] Helin, Henrik O., et al. "Free digital image analysis software helps to resolve equivocal scores in CERBB2 immunohistochemistry." Virchows Archiv 468.2, 191-198, 2016.
[8] Pezoa, Raquel, et al. "Segmentation of HER2 protein overexpression in immunohistochemically stained breast cancer images using Support Vector Machines." Journal of Physics: Conference Series. Cilt. 762. SayÕ. 1. IOP Publishing, 2016.
[9] Tang, Jinshan, et al. "Computer-aided detection and diagnosis of breast cancer with mammography: recent advances." IEEE Transactions on Information Technology in Biomedicine 13.2, 236-251, 2009.
[10] Cireúan, Dan C., et al. "Mitosis detection in breast cancer histology images with deep neural networks." International Conference on Medical Image Computing and Computer-assisted Intervention. Springer Berlin Heidelberg, 2013.
[11] Wang, Dayong, et al. "Deep learning for identifying metastatic breast cancer." arXiv preprint arXiv:1606.05718, 2016.
[12] Geçer, BarÕú. Detection and classification of breast cancer in whole slide histopathology images using deep convolutional networks. Diss. Bilkent University, 2016.
[13] Ruifrok, Arnout C., and Dennis A. Johnston. "Quantification of histochemical staining by color deconvolution." Analytical and quantitative cytology and histology 23.4, 291-299, 2001.
[14] KabakçÕ, K. A., Çapar, A., Töreyin, B. U., Akkoç, M., Borazan, O., Türkmen, ø ., Durak Ata, L., “Mikroskobik Floresan In Situ Hibridizasyon (FISH) Görüntülerde Çoklu Seviye Eúikleme TabanlÕ Bölütleme Yöntemi”, 24. IEEE Sinyal øúleme ve øletiúim UygulamalarÕ KurultayÕ, 2016.
[15] Schmidhuber, Jürgen. "Deep learning in neural networks: An overview." Neural networks 61, 85-117, 2015.
[16] Jia, Yangqing, et al. "Caffe: Convolutional architecture for fast feature embedding." Proceedings of the 22nd ACM international conference on Multimedia. ACM, 2014.
[17] Yue-Hei Ng, Joe, et al. "Beyond short snippets: Deep networks for video classification." Proceedings of the IEEE conference on computer vision and pattern recognition, 2015.
[18] Chen, Liang-Chieh, et al. "Semantic image segmentation with deep convolutional nets and fully connected crfs." arXiv preprint arXiv:1412.7062, 2014.
[19] Fan, Rong-En, et al. "LIBLINEAR: A library for large linear classification." Journal of machine learning research, 1871-1874, 2008. [20] Gong, Yunchao, et al. "Deep convolutional ranking for multilabel image
annotation." arXiv preprint arXiv:1312.4894, 2013.
[21] Wu, Yichao, and Yufeng Liu. "Robust truncated hinge loss support vector machines." Journal of the American Statistical Association 102.479, 974-983, 2007.
[22] Joachims, Thorsten. Making large-scale SVM learning practical. SayÕ. 1998, 28. Technical Report, SFB 475: Komplexitätsreduktion in Multivariaten Datenstrukturen, Universität Dortmund, 1998.
![Tablo 1. Veri kümesindeki her bir skor türü için kesilen bazÕ hücre parçalarÕ [6].](https://thumb-eu.123doks.com/thumbv2/9libnet/5437725.104175/2.892.103.407.78.368/tablo-veri-kümesindeki-türü-kesilen-bazõ-hücre-parçalarõ.webp)